No Outono, os grilos geralmente exibem canções de chilrear no país temperado do leste asiático do Japão. Enquanto o grilo de campo africano Gryllus bimaculatus é originário de países tropicais, ele é um animal modelo emergente globalmente devido a sua capacidade de regenerar as pernas amputadas durante a ninfa e seu modo de desenvolvimento (banda germinativa curta) (Mito e Noji, 2008).
Muitos organismos vivos no reino animal são capazes de regenerar suas partes do corpo após lesões. Exemplos de partes do corpo que podem ser regredidas incluem as lentes e a cauda dos anfíbios, a cabeça dos planários, e o coração dos peixes. Em contraste, há muito se supõe que os humanos não podem restaurar partes do corpo perdidas, exceto por tecidos particulares, incluindo a epiderme, o fígado, e a superfície ovariana após a ovulação. Portanto, é importante elucidar os mecanismos moleculares envolvidos nos processos de regeneração usando modelos animais capazes de regenerar partes do corpo para posterior aplicação em órgãos e tecidos humanos não regenerativos.
Nos últimos 2 anos, estudos genômicos comparativos de duas espécies de planários com diferentes habilidades regenerativas levaram à regeneração bem sucedida das cabeças, reduzindo a atividade da beta-catenina a partir de fragmentos de cauda não regenerativa (Umesono et al., 2013). Estudos de vertebrados com capacidade de restaurar membros, incluindo tritões, sapos e salamandras, demonstraram que a regeneração dos membros ocorre de forma gradual. O processo de regeneração de membros é dividido em pelo menos três fases: cicatrização, desdiferenciação e redesenvolvimento, com a fase de redesenvolvimento imitando o desenvolvimento embrionário (Endo et al., 2004).
A perna de grilo é composta de seis segmentos que estão dispostos ao longo do eixo proximo-distal (PD): coxa, trocânter, fêmur, tíbia, tarso e garra (Figura 1). 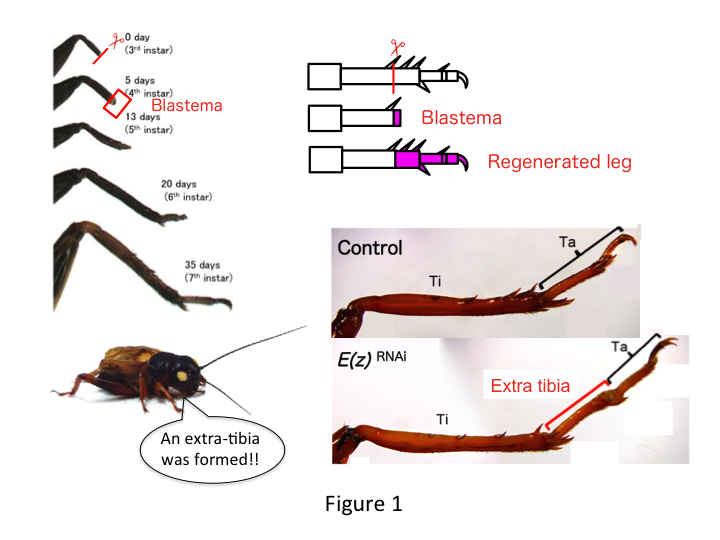 O tarso é ainda subdividido em três átomos de tarso. Quando a tíbia da ninfa estrela do terceiro é amputada, a perna se regenera e recupera seu tamanho alométrico e forma adequada pelo sexto instar (ou seja, dentro de 20 dias da amputação), sendo restaurada ao tamanho e forma adultos quase normais. Logo após a cura, a blastema (um pool de células que proliferam) desenvolve-se na região distal da perna amputada. As células do blastema proliferam e formam as estruturas ausentes por processos intercalares entre a região mais distal e a parte restante da perna (French et al., 1976).
O tarso é ainda subdividido em três átomos de tarso. Quando a tíbia da ninfa estrela do terceiro é amputada, a perna se regenera e recupera seu tamanho alométrico e forma adequada pelo sexto instar (ou seja, dentro de 20 dias da amputação), sendo restaurada ao tamanho e forma adultos quase normais. Logo após a cura, a blastema (um pool de células que proliferam) desenvolve-se na região distal da perna amputada. As células do blastema proliferam e formam as estruturas ausentes por processos intercalares entre a região mais distal e a parte restante da perna (French et al., 1976).
Anteriormente, realizamos análise comparativa de transcriptoma de pernas regeneradas e amputadas normais de grilos para perfilar a expressão do mRNA associado à regeneração da perna (Bando et al., 2013). Focalizamos primeiro a upregulação dos genes da via Jak/Stat, que estão ligados ao sistema imunológico. A interferência do RNAi (RNAi) dos genes nesta via perturbou profundamente a regeneração das pernas. Em contraste, o RNAi contra Socs, um supressor de sinalização de citocinas, causou o alongamento das pernas. Experimentos adicionais mostraram que a via Jak/Stat promove a proliferação celular a jusante da via Ds/Fat.
Subseqüentemente, nós investigamos a regulação epigenética durante a regeneração da perna de críquete. Tetsuya Bando, um investigador sênior do nosso grupo, identificou um gene para a histona H3 lisina 27 (H3K27) metiltransferase, E(z), e um gene para a histona H3K27 demetilase, Utx, em G. bimaculatus. A clonagem dos genes Gryllus é agora um processo simples devido à informação disponível sobre o genoma do críquete (Mito e Noji, comunicação pessoal). A metilação da histona H3K27 por E(z) reprime a expressão dos genes alvo através do recrutamento de proteínas do grupo Polycomb. Por outro lado, a desmetilação da histona trimetilada H3K27 por Utx promove a expressão dos genes. Tetsuya descobriu que a transcrição dos genes E(z) e Utx é upregulada nas células blastemáticas das pernas amputadas (Bando et al., 2013). A hibridação in situ verificou que ambos os genes são transcritos ubíquamente nas pernas regeneradoras dos grilos, e que ambos os genes são expressos em embriões em desenvolvimento (Hamada et al., 2015). Imunostaining on the amputated tiny legs after RNAi by Yoshimasa Hamada (a PhD student) confirmed that E(z) and Utx contribute to the methylation and demethylation at histone H3K27me3, respectively, during leg regeneration.
However, Yoshimasa unexpectedly found that the extra leg segment is formed after RNAi against E(z) (Figure 1). Inicialmente, não foi possível determinar a identidade do segmento da perna. Morfologicamente, o segmento de perna parecia ser uma tíbia, pois tinha espinhos e esporas característicos de uma tíbia autêntica. Nossa hipótese inicial era que os fenótipos após o RNAi poderiam depender do local da amputação na tíbia. Entretanto, mesmo quando uma perna é amputada na parte distal do fêmur, emerge o segmento extra tipo tíbia. A formação do padrão ao longo dos eixos antero-posterior e dorso-ventral permaneceu inalterada, exceto ao longo do eixo PD. Examinamos então se o local da amputação ao longo do eixo da DP na tíbia influenciou a severidade fenotípica. A extra-tíbia que se formou tornou-se mais longa quanto mais proximais os locais de amputação na tíbia (Figura 1). Por outro lado, RNAi contra Utx resultou na perda da formação articular entre o tarsomero 1 (Ta1) e Ta2 (Figura 2). 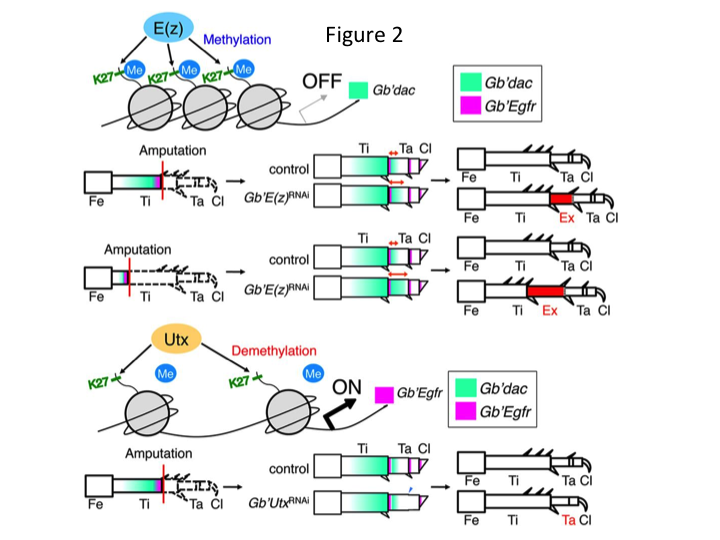 A hibridação in situ mostrou que a expressão dos genes de padrões de perna alterados ao longo do eixo PD. Especificamente, o domínio da expressão do dachshund (dac) expandiu-se em pernas regeneradoras E(z)RNAi, enquanto a expressão do Egfr diminuiu em pernas UtxRNAi. Portanto, E(z) pode reprimir a expressão dac durante a regeneração normal das pernas, enquanto que Utx induz a expressão Egfr.
A hibridação in situ mostrou que a expressão dos genes de padrões de perna alterados ao longo do eixo PD. Especificamente, o domínio da expressão do dachshund (dac) expandiu-se em pernas regeneradoras E(z)RNAi, enquanto a expressão do Egfr diminuiu em pernas UtxRNAi. Portanto, E(z) pode reprimir a expressão dac durante a regeneração normal das pernas, enquanto que Utx induz a expressão Egfr.
dac codifica um co-repressor transcripcional que é categorizado em genes de leg gap. dac produz valores posicionais brutos ao longo do eixo PD da perna e medeia a formação da tíbia distal e Ta1 (o tarsomere proximal) durante a regeneração da perna de críquete (o domínio da expressão dac é mostrado em verde na Figura 2) (Ishimaru et al., 2015). Especificamente, o dac promove a proliferação da célula tibial. Portanto, porque RNAi contra E(z) upregula o dac, a expressão E(z) nas células blastema pode suprimir a superproliferação blastemal através da repressão da expressão extra do dac.
Esta informação levanta a questão de como E(z) regula especificamente a expressão do dac. Além disso, qual é o mecanismo que determina os genes alvo de E(z)? E(z) pertence ao complexo repressivo Polycomb 2 (PRC2), que é um dos três complexos do grupo Polycomb (PcG) (Schuettengruber et al., 2007). Durante a embriogênese do cricket, E(z) reprime a expansão anterior da expressão do gene Hox e fornece identidade própria em embriões (Matsuoka et al., 2015). Esta informação indica que os genes alvo de E(z) diferem dependendo do contexto celular. Uma proteína de ligação ao DNA, Pleiohomeotic (Pho), juntamente com outros fatores, liga-se aos elementos de resposta Polycomb (PRE) dos genes alvo, após o que E(z) trimetilatos histone H3K27. Embora os PREs só tenham sido identificados em Drosophila, a meta-análise dos genes alvo putativos para as proteínas PcG mostrou que muitos dos genes alvo são comuns à mosca, ao rato e aos humanos. dac e Egfr estão incluídos entre estes genes (Schuettengruber et al., 2007). Assim, a região reguladora do gene do dac de críquete provavelmente contém PREs, através dos quais E(z) regula epigenicamente a expressão do dac durante a regeneração da perna de críquete (Figura 2). Pesquisas em andamento estão focadas na caracterização das funções do gene Pho e outros genes do complexo PcG e modificadores epigenéticos durante a regeneração da perna de Gryllus.
Finalmente, por que o E(z) RNAi causa a formação de extra-tibíase? Um cenário hipotético é que quando a tíbia é amputada na posição proximal onde a expressão dac é baixa, a expressão Utx (que domina a expressão E(z)) permite a expressão dac (Figura 3a) para restaurar a tíbia. 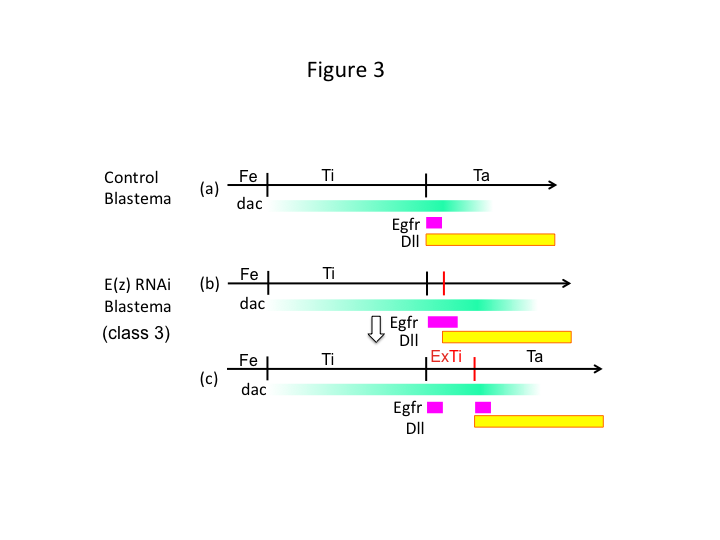 Assim, estes modificadores de histone sentem os valores posicionais ao longo do eixo PD do local da amputação, e afinam o nível de expressão dos genes de patterning das pernas, como dac. No caso do E(z) RNAi imediatamente antes da amputação proximal, a expressão intensa do dac é induzida e se expande na perna regeneradora (Figura 3b). A expressão sem distal (Dll), que é outro gene de distal que especifica o domínio distal da perna (Angelini e Kaufman, 2005), pode mudar mais distalmente dependendo da expressão expandida do dac (Figura 3b). Assim, o domínio Egfr-expressor pode ser separado em duas partes onde (1) a expressão Dll é baixa e (2) Dll é alta. A extra-tibia provavelmente se forma entre os dois domínios diferentes do Egfr-expressor intercalando a proliferação celular e o patterning (Figura 3c).
Assim, estes modificadores de histone sentem os valores posicionais ao longo do eixo PD do local da amputação, e afinam o nível de expressão dos genes de patterning das pernas, como dac. No caso do E(z) RNAi imediatamente antes da amputação proximal, a expressão intensa do dac é induzida e se expande na perna regeneradora (Figura 3b). A expressão sem distal (Dll), que é outro gene de distal que especifica o domínio distal da perna (Angelini e Kaufman, 2005), pode mudar mais distalmente dependendo da expressão expandida do dac (Figura 3b). Assim, o domínio Egfr-expressor pode ser separado em duas partes onde (1) a expressão Dll é baixa e (2) Dll é alta. A extra-tibia provavelmente se forma entre os dois domínios diferentes do Egfr-expressor intercalando a proliferação celular e o patterning (Figura 3c).
Nosso objetivo é elucidar os planos para “fazer uma perna regenerada” usando este modelo atrativo de insetos hemimetabolos. Espera-se que os planos esclareçam como é determinado o número de segmentos de perna. As nossas observações marcantes sobre o RNAi contra E(z) que conduzem à “formação de tíbia extra” representam um passo importante para elucidar este processo.
- Mito, T. e Noji, S. (2008). O Gryllus bimaculatus de críquete de dois pontos: Um modelo emergente para Estudos de Desenvolvimento e Regeneração. Cold Spring Harb Protoc, 331-346.
- Umesono, Y., Tasaki, J., Nishimura, Y., Hrouda, M., Kawaguchi, E., Yazawa, S., Nishimura, O., Hosoda, K., Inoue, T. e Agata, K. (2013). A lógica molecular para a regeneração planetária ao longo do eixo anterior-posterior. Nature 500, 73-76.
- Endo, T., Bryant, S. V. e Gardiner, D. M. (2004). Um sistema modelo passo a passo para a regeneração dos membros. Dev Biol 270, 135-145.
- French, V., Bryant, P. J. e Bryant, S. V. (1976). Regulação de padrões em campos epimórficos. Science 193, 969-981.
- Bando, T., Ishimaru, Y., Kida, T., Hamada, Y., Matsuoka, Y., Nakamura, T., Ohuchi, H., Noji, S. e Mito, T. (2013). A análise dos dados do RNA-Seq revela o envolvimento da sinalização JAK/STAT durante a regeneração das pernas no grilo Gryllus bimaculatus. Desenvolvimento 140, 959-964.
- Hamada, Y., Bando, T., Nakamura, T., Ishimaru, Y., Mito, T., Noji, S., Tomioka, K. e Ohuchi, H. (2015). A regeneração das pernas é regulada epigenicamente pela metilação do histone H3K27 no grilo Gryllus bimaculatus. Desenvolvimento 142, 2916-2927.
- Ishimaru, Y., Nakamura, T., Bando, T., Matsuoka, Y., Ohuchi, H., Noji, S. e Mito, T. (2015). Envolvimento de Teckel e Distal-less na formação do padrão distal da perna de grilo durante a regeneração. Sci Rep 5, 8387.
- Schuettengruber, B., Chourrout, D., Vervoort, M., Leblanc, B. e Cavalli, G. (2007). Regulação do genoma por polycomb e proteínas tritórax. Célula 128, 735-745.
- Matsuoka, Y., Bando, T., Watanabe, T., Ishimaru, Y., Noji, S., Popadić, A. e Mito, T. (2015). Os insetos de germes curtos utilizam tanto o modo ancestral como o modo derivado do silenciamento epigenético dos genes Hox, mediado pelo grupo Polycomb. Biol Open 4, 702-709.
- Angelini,R. e Kaufman, T. C. (2005). Apêndices de insectos e ontogenética comparativa. Dev Biol 286, 57-77.
Partilhar
- Edição:*
- O seu nome:*
- O seu e-mail:*
- Detalhes:*
Enviar Relatório:*5713>
 (1 voto)
(1 voto)
Tags: Cromatina e epigenética, cricket, regeneração
Categorias: Pesquisa