En otoño, los grillos suelen exhibir cantos de gorjeo en el país templado de Asia oriental, Japón. Aunque el grillo de campo africano Gryllus bimaculatus es originario de países tropicales, es un animal modelo emergente a nivel mundial por su capacidad de regenerar las patas amputadas durante la ninfa y su modo de desarrollo (banda germinal corta) (Mito y Noji, 2008).
Muchos organismos vivos del reino animal son capaces de regenerar sus partes del cuerpo tras una lesión. Algunos ejemplos de partes del cuerpo que pueden volver a crecer son el cristalino y la cola de los anfibios, la cabeza de los planarios y el corazón de los peces. En cambio, durante mucho tiempo se ha asumido que los seres humanos no pueden restaurar las partes del cuerpo perdidas, excepto determinados tejidos, como la epidermis, el hígado y la superficie de los ovarios tras la ovulación. Por lo tanto, es importante dilucidar los mecanismos moleculares implicados en los procesos de regeneración utilizando modelos animales capaces de regenerar partes del cuerpo para su posterior aplicación en órganos y tejidos humanos no regenerativos.
En los últimos 2 años, los estudios genómicos comparativos de dos especies de planarias con diferentes capacidades regenerativas condujeron a la regeneración exitosa de cabezas mediante la reducción de la actividad de la beta-catenina a partir de fragmentos de cola que de otro modo no se regenerarían (Umesono et al., 2013). Los estudios de los vertebrados con capacidad para restaurar las extremidades, incluidos los tritones, las ranas y las salamandras, han demostrado que la regeneración de las extremidades se produce de forma escalonada. El proceso de regeneración de las extremidades se divide en al menos tres fases: cicatrización de la herida, desdiferenciación y redesarrollo, siendo la fase de redesarrollo la que imita el desarrollo embrionario (Endo et al., 2004).
La pata del grillo está compuesta por seis segmentos que se disponen a lo largo del eje proximodistal (PD): coxa, trocánter, fémur, tibia, tarso y garra (Figura 1). 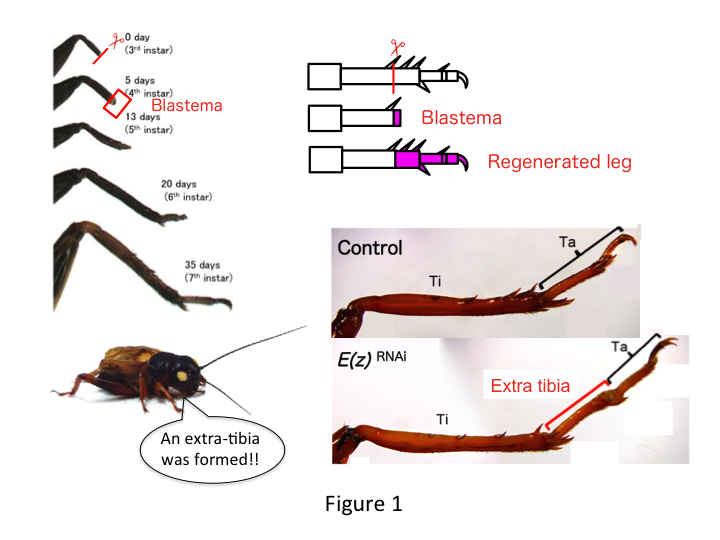 El tarso se subdivide además en tres tarsómeros. Cuando se amputa la tibia de la ninfa del tercer estadio, la pata se regenera y recupera su tamaño alométrico y su forma adecuada en el sexto estadio (es decir, a los 20 días de la amputación), recuperando un tamaño y una forma casi normales de adulto. Poco después de la curación, el blastema (un conjunto de células que proliferan) se desarrolla en la región distal de la pata amputada. Las células del blastema proliferan y forman las estructuras que faltan mediante procesos intercalares entre la región más distal y la parte restante de la pata (French et al., 1976).
El tarso se subdivide además en tres tarsómeros. Cuando se amputa la tibia de la ninfa del tercer estadio, la pata se regenera y recupera su tamaño alométrico y su forma adecuada en el sexto estadio (es decir, a los 20 días de la amputación), recuperando un tamaño y una forma casi normales de adulto. Poco después de la curación, el blastema (un conjunto de células que proliferan) se desarrolla en la región distal de la pata amputada. Las células del blastema proliferan y forman las estructuras que faltan mediante procesos intercalares entre la región más distal y la parte restante de la pata (French et al., 1976).
Previamente, realizamos un análisis comparativo del transcriptoma de patas regeneradas y normales amputadas de grillos para perfilar la expresión de ARNm asociada a la regeneración de la pata (Bando et al., 2013). En primer lugar, nos centramos en la regulación al alza de los genes de la vía Jak/Stat, que están vinculados al sistema inmunitario. La interferencia de ARN (ARNi) de los genes de esta vía perturbó completamente la regeneración de la pata. En cambio, la RNAi contra Socs, un supresor de la señalización de citoquinas, provocó la elongación de las piernas. Otros experimentos demostraron que la vía Jak/Stat promueve la proliferación de las células en la parte inferior de la vía Ds/Grasa.
A continuación, investigamos la regulación epigenética durante la regeneración de las patas de los grillos. Tetsuya Bando, investigador principal de nuestro grupo, identificó un gen de la histona H3 lisina 27 (H3K27) metiltransferasa, E(z), y un gen de la histona H3K27 desmetilasa, Utx, en G. bimaculatus. La clonación de genes de Gryllus es ahora un proceso sencillo gracias a la información disponible sobre el genoma del grillo (Mito y Noji, comunicación personal). La metilación de la histona H3K27 por parte de E(z) reprime la expresión de los genes objetivo reclutando a las proteínas del grupo Polycomb. Por el contrario, la desmetilación de la histona H3K27 trimetilada por Utx promueve la expresión de los genes. Tetsuya descubrió que la transcripción de los genes E(z) y Utx está regulada al alza en las células del blastema de las piernas amputadas (Bando et al., 2013). La hibridación in situ verificó que ambos genes se transcriben de forma ubicua en las patas regeneradas de los grillos, y que ambos genes se expresan en los embriones en desarrollo (Hamada et al., 2015). La inmunotinción en las patas diminutas amputadas después de RNAi por Yoshimasa Hamada (estudiante de doctorado) confirmó que E(z) y Utx contribuyen a la metilación y desmetilación en la histona H3K27me3, respectivamente, durante la regeneración de las patas.
Sin embargo, Yoshimasa encontró inesperadamente que el segmento extra de la pata se forma después de RNAi contra E(z) (Figura 1). Inicialmente, no pudimos determinar la identidad del segmento de la pata. Morfológicamente, el segmento de la pata parecía ser una tibia, porque tenía espinas y espolones característicos de una tibia auténtica. Nuestra hipótesis inicial era que los fenotipos tras el ARNi podrían depender del lugar de amputación en la tibia. Sin embargo, incluso cuando se amputa una pata en la parte distal del fémur, surge el segmento extra tibial. La formación del patrón a lo largo de los ejes antero-posterior y dorso-ventral se mantuvo sin cambios, excepto a lo largo del eje PD. A continuación, examinamos si el lugar de la amputación a lo largo del eje PD en la tibia influía en la gravedad del fenotipo. La extra-tibia que se formó se hizo más larga cuanto más proximales eran los sitios de amputación en la tibia (Figura 1). Por el contrario, el ARNi contra Utx dio lugar a la pérdida de la formación de la articulación entre el tarsómero 1 (Ta1) y Ta2 (Figura 2). 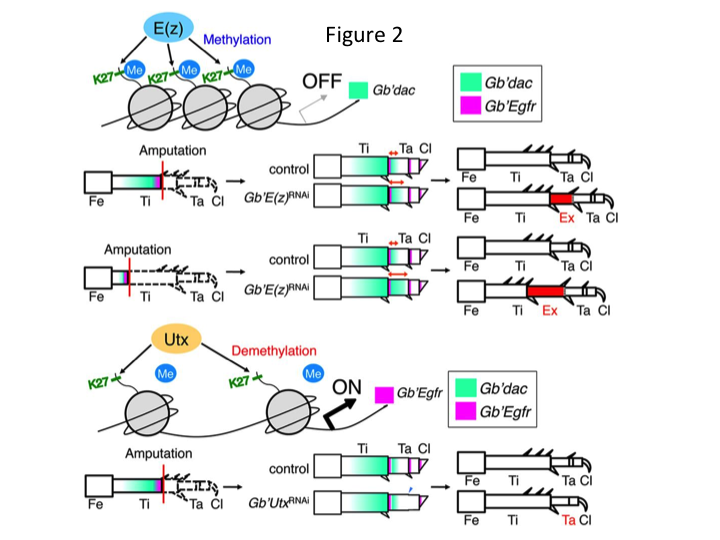 La hibridación in situ mostró que la expresión de los genes del patrón de la pata se alteraba a lo largo del eje de la PD. En concreto, el dominio de la expresión de dachshund (dac) se expandió en las patas regeneradas por E(z)RNAi, mientras que la expresión de Egfr disminuyó en las patas UtxRNAi. Por lo tanto, E(z) puede reprimir la expresión de dac durante la regeneración normal de las patas, mientras que Utx induce la expresión de Egfr.
La hibridación in situ mostró que la expresión de los genes del patrón de la pata se alteraba a lo largo del eje de la PD. En concreto, el dominio de la expresión de dachshund (dac) se expandió en las patas regeneradas por E(z)RNAi, mientras que la expresión de Egfr disminuyó en las patas UtxRNAi. Por lo tanto, E(z) puede reprimir la expresión de dac durante la regeneración normal de las patas, mientras que Utx induce la expresión de Egfr.
dac codifica un correpresor transcripcional que está categorizado en los genes de la brecha de la pata. dac produce valores posicionales brutos a lo largo del eje de la pata y media la formación de la tibia distal y Ta1 (el tarsómero proximal) durante la regeneración de la pata de grillo (el dominio de expresión de dac se muestra en verde en la Figura 2) (Ishimaru et al., 2015). En concreto, dac promueve la proliferación de las células tibiales. Por lo tanto, dado que el ARNi contra E(z) regula al alza dac, la expresión de E(z) en las células del blastema puede suprimir la sobreproliferación blastemal reprimiendo la expresión extra de dac.
Esta información plantea la cuestión de cómo E(z) regula específicamente la expresión de dac. Además, ¿cuál es el mecanismo que determina los genes diana de E(z)? E(z) pertenece al complejo represivo Polycomb 2 (PRC2), que es uno de los tres complejos del grupo Polycomb (PcG) (Schuettengruber et al., 2007). Durante la embriogénesis del grillo, E(z) reprime la expansión anterior de la expresión de los genes Hox y proporciona una identidad adecuada en los embriones (Matsuoka et al., 2015). Esta información indica que los genes objetivo de E(z) difieren según el contexto celular. Una proteína de unión al ADN, Pleiohomeotic (Pho), junto con otros factores, se une a los elementos de respuesta Polycomb (PRE) de los genes diana, tras lo cual E(z) trimetila la histona H3K27. Aunque los PREs sólo se han identificado en Drosophila, el meta-análisis de los genes diana putativos para las proteínas PcG ha mostrado que muchos de los genes diana son comunes a la mosca, el ratón y los humanos. dac y Egfr están incluidos entre estos genes (Schuettengruber et al., 2007). Así pues, la región reguladora del gen dac del grillo contiene probablemente PREs, a través de los cuales E(z) regula epigenéticamente la expresión de dac durante la regeneración de la pata del grillo (Figura 2). La investigación en curso se centra en la caracterización de las funciones del gen Pho y otros genes del complejo PcG y modificadores epigenéticos durante la regeneración de la pata de Gryllus.
Por último, ¿por qué el ARNi de E(z) causa la formación de extra-tibia? Un escenario hipotético es que cuando la tibia se amputa en la posición proximal, donde la expresión de dac es baja, la expresión de Utx (que domina la expresión de E(z)) permite la expresión de dac (Figura 3a) para restaurar la tibia. 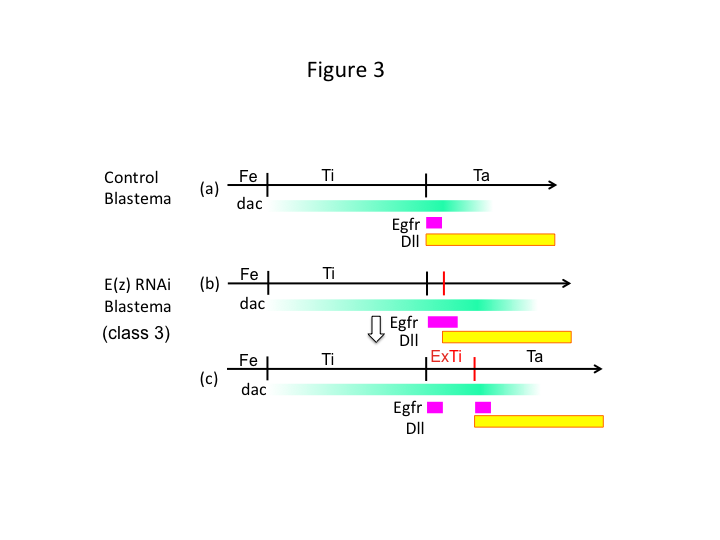 Así, estos modificadores de histonas detectan los valores posicionales a lo largo del eje de la DP del lugar de la amputación, y ajustan el nivel de expresión de los genes del patrón de la pierna, como dac. En el caso del ARNi de E(z) justo antes de la amputación proximal, se induce una intensa expresión de dac que se expande en la pierna en regeneración (Figura 3b). La expresión de Distal-less (Dll), que es otro gen de la pata que especifica el dominio distal de la pata (Angelini y Kaufman, 2005), puede desplazarse más distalmente en función de la expresión expandida de dac (Figura 3b). Así, el dominio que expresa Egfr puede estar separado en dos partes donde (1) la expresión de Dll es baja y (2) Dll es alta. La extra-tibia probablemente se forma entre los dos dominios diferentes que expresan Egfr mediante la proliferación celular intercalada y el modelado (Figura 3c).
Así, estos modificadores de histonas detectan los valores posicionales a lo largo del eje de la DP del lugar de la amputación, y ajustan el nivel de expresión de los genes del patrón de la pierna, como dac. En el caso del ARNi de E(z) justo antes de la amputación proximal, se induce una intensa expresión de dac que se expande en la pierna en regeneración (Figura 3b). La expresión de Distal-less (Dll), que es otro gen de la pata que especifica el dominio distal de la pata (Angelini y Kaufman, 2005), puede desplazarse más distalmente en función de la expresión expandida de dac (Figura 3b). Así, el dominio que expresa Egfr puede estar separado en dos partes donde (1) la expresión de Dll es baja y (2) Dll es alta. La extra-tibia probablemente se forma entre los dos dominios diferentes que expresan Egfr mediante la proliferación celular intercalada y el modelado (Figura 3c).
Nuestro objetivo es dilucidar los planos para «hacer una pierna regenerada» utilizando este atractivo modelo de insecto hemimetábolo. Se espera que los planos aclaren cómo se determina el número de segmentos de la pata. Nuestras sorprendentes observaciones sobre el ARNi contra E(z) que conduce a la «formación de tibias adicionales» representan un paso importante hacia la elucidación de este proceso.
- Mito, T. y Noji, S. (2008). El grillo de dos manchas Gryllus bimaculatus: Un modelo emergente para estudios de desarrollo y regeneración. Cold Spring Harb Protoc, 331-346.
- Umesono, Y., Tasaki, J., Nishimura, Y., Hrouda, M., Kawaguchi, E., Yazawa, S., Nishimura, O., Hosoda, K., Inoue, T. y Agata, K. (2013). La lógica molecular para la regeneración de las planarias a lo largo del eje anterior-posterior. Nature 500, 73-76.
- Endo, T., Bryant, S. V. y Gardiner, D. M. (2004). Un sistema modelo por etapas para la regeneración de las extremidades. Dev Biol 270, 135-145.
- French, V., Bryant, P. J. y Bryant, S. V. (1976). Regulación de patrones en campos epimórficos. Science 193, 969-981.
- Bando, T., Ishimaru, Y., Kida, T., Hamada, Y., Matsuoka, Y., Nakamura, T., Ohuchi, H., Noji, S. y Mito, T. (2013). Analysis of RNA-Seq data reveals involvement of JAK/STAT signalling during leg regeneration in the cricket Gryllus bimaculatus. Development 140, 959-964.
- Hamada, Y., Bando, T., Nakamura, T., Ishimaru, Y., Mito, T., Noji, S., Tomioka, K. y Ohuchi, H. (2015). La regeneración de las patas está regulada epigenéticamente por la metilación de la histona H3K27 en el grillo Gryllus bimaculatus. Development 142, 2916-2927.
- Ishimaru, Y., Nakamura, T., Bando, T., Matsuoka, Y., Ohuchi, H., Noji, S. y Mito, T. (2015). Implicación de dachshund y Distal-less en la formación del patrón distal de la pata de grillo durante la regeneración. Sci Rep 5, 8387.
- Schuettengruber, B., Chourrout, D., Vervoort, M., Leblanc, B. y Cavalli, G. (2007). Regulación del genoma por las proteínas polycomb y trithorax. Cell 128, 735-745.
- Matsuoka, Y., Bando, T., Watanabe, T., Ishimaru, Y., Noji, S., Popadić, A. y Mito, T. (2015). Los insectos de germen corto utilizan tanto el modo ancestral como el derivado del silenciamiento epigenético mediado por el grupo Polycomb de los genes Hox. Biol Open 4, 702-709.
- Angelini,R. y Kaufman, T. C. (2005). Apéndices de insectos y ontogenética comparativa. Dev Biol 286, 57-77.
Compartir
- Tema:*
- Su nombre:*
- Su correo electrónico:*
- Detalles:*
Enviar informe
 (1 votos)
(1 votos)
Etiquetas: Cromatina y epigenética, grillo, regeneración
Categorías: Investigación