W jesieni, świerszcze ogólnie wykazują świergotanie piosenek w umiarkowanej Azji Wschodniej kraju Japonii. Podczas gdy afrykański świerszcz polny Gryllus bimaculatus pochodzi z krajów tropikalnych, jest to wschodzące zwierzę modelowe na całym świecie ze względu na jego zdolność do regeneracji amputowanych nóg podczas nimfy i jego tryb rozwoju (krótkie pasmo zarodkowe) (Mito i Noji, 2008).
Wiele żywych organizmów w królestwie zwierząt jest w stanie odrastać ich części ciała po urazie. Przykłady części ciała, które mogą być odrastające obejmują soczewki i ogon płazów, głowy planariów i serca ryb. W przeciwieństwie do tego, od dawna zakłada się, że człowiek nie może odtworzyć utraconych części ciała, z wyjątkiem niektórych tkanek, takich jak naskórek, wątroba i powierzchnia jajnika po owulacji. Dlatego ważne jest, aby wyjaśnić mechanizmy molekularne zaangażowane w procesy regeneracji przy użyciu modeli zwierzęcych, które są zdolne do regeneracji części ciała w celu późniejszego zastosowania w nieregenerujących się ludzkich narządach i tkankach.
W ciągu ostatnich 2 lat, porównawcze badania genomiczne dwóch gatunków planarian o różnych zdolnościach regeneracyjnych doprowadziły do udanej regeneracji głów poprzez zmniejszenie aktywności beta-kateniny z inaczej nieregenerujących się fragmentów ogona (Umesono i in., 2013). Badania kręgowców posiadających zdolność do odbudowy kończyn, w tym traszek, żab i salamander, wykazały, że regeneracja kończyn zachodzi w sposób stopniowy. Proces regeneracji kończyn dzieli się na co najmniej trzy fazy: gojenie się ran, dedifferentację i ponowny rozwój, przy czym faza ponownego rozwoju naśladuje rozwój embrionalny (Endo i in., 2004).
Noga świerszcza składa się z sześciu segmentów, które są ułożone wzdłuż osi proksymalno-dystalnej (PD): coxa, trochanter, femur, tibia, tarsus i claw (Rycina 1).
Poprzednio przeprowadziliśmy porównawczą analizę transkryptomu regenerujących i normalnych amputowanych nóg świerszczy w celu profilowania ekspresji mRNA związanej z regeneracją nóg (Bando i in., 2013). W pierwszej kolejności skupiliśmy się na upregulacji genów szlaku Jak/Stat, które są związane z układem odpornościowym. Interferencja RNA (RNAi) genów w tym szlaku gruntownie zaburzyła regenerację nóg. Z kolei RNAi przeciwko Socs, supresorowi sygnalizacji cytokinowej, powodowało wydłużanie się nóg. Dodatkowe eksperymenty wykazały, że szlak Jak/Stat promuje proliferację komórek poniżej szlaku Ds/Fat.
Później zbadaliśmy regulację epigenetyczną podczas regeneracji nóg świerszcza. Tetsuya Bando, starszy badacz w naszej grupie, zidentyfikował jeden gen dla metylotransferazy histonu H3 lizyny 27 (H3K27), E(z), i jeden gen dla demetylazy histonu H3K27, Utx, w G. bimaculatus. Klonowanie genów Gryllus jest obecnie prostym procesem ze względu na dostępność informacji o genomie świerszcza (Mito i Noji, komunikacja osobista). Metylacja histonu H3K27 przez E(z) hamuje ekspresję genów docelowych poprzez rekrutację białek grupy Polycomb. I odwrotnie, demetylacja trimetylowanego histonu H3K27 przez Utx promuje ekspresję genów. Tetsuya odkrył, że transkrypcja zarówno genów E(z), jak i Utx jest podwyższona w komórkach blastemy amputowanych kończyn (Bando i in., 2013). Hybrydyzacja in situ zweryfikowała, że oba geny są wszechobecnie transkrybowane w regenerujących się nogach świerszczy, a także, że ulegają ekspresji w rozwijających się zarodkach (Hamada i in., 2015). Immunostaining na amputowanych malutkich nogach po RNAi przez Yoshimasę Hamadę (doktoranta) potwierdził, że E(z) i Utx przyczyniają się do metylacji i demetylacji w histonie H3K27me3, odpowiednio, podczas regeneracji nóg.
Jednakże Yoshimasa nieoczekiwanie stwierdził, że dodatkowy segment nogi powstaje po RNAi przeciwko E(z) (Figura 1). Początkowo nie byliśmy w stanie określić tożsamości segmentu nogi. Morfologicznie, segment nogi wydawał się być piszczelą, ponieważ miał kolce i ostrogi charakterystyczne dla autentycznej piszczeli. Naszą początkową hipotezą było to, że fenotypy po RNAi mogą zależeć od miejsca amputacji w piszczeli. Jednakże, nawet gdy noga jest amputowana w dystalnej części kości udowej, pojawia się dodatkowy segment podobny do piszczeli. Formowanie wzorów wzdłuż osi przednio-tylnej i grzbietowo-wewnętrznej pozostało niezmienione, z wyjątkiem osi PD. Następnie sprawdziliśmy, czy miejsce amputacji wzdłuż osi PD w kości piszczelowej wpływa na ciężkość fenotypu. Tworząca się kość piszczelowa stawała się tym dłuższa, im bardziej proksymalne były miejsca amputacji na kości piszczelowej (Rysunek 1). I odwrotnie, RNAi przeciwko Utx spowodowało utratę tworzenia się stawów między stępem 1 (Ta1) i Ta2 (Figura 2).
dac koduje transkrypcyjny ko-represor, który jest skategoryzowany w genach przerwy w nodze. dac wytwarza surowe wartości pozycyjne wzdłuż osi PD nogi i pośredniczy w tworzeniu dystalnej kości piszczelowej i Ta1 (proksymalnego stępu) podczas regeneracji nogi świerszcza (domena ekspresji dac jest pokazana na zielono na rycinie 2) (Ishimaru i in., 2015). Konkretnie, dac promuje proliferację komórek piszczelowych. Dlatego, ponieważ RNAi przeciwko E(z) podwyższa ekspresję dac, ekspresja E(z) w komórkach blastemy może tłumić nadproliferację blastemali poprzez represję dodatkowej ekspresji dac.
Ta informacja rodzi pytanie, w jaki sposób E(z) specyficznie reguluje ekspresję dac. Co więcej, jaki jest mechanizm, który determinuje docelowe geny E(z)? E(z) należy do kompleksu represyjnego Polycomb 2 (PRC2), który jest jednym z trzech kompleksów grupy Polycomb (PcG) (Schuettengruber i in., 2007). Podczas embriogenezy świerszcza, E(z) represjonuje przednią ekspansję ekspresji genów Hox i zapewnia prawidłową tożsamość zarodków (Matsuoka i in., 2015). Informacje te wskazują, że geny docelowe E(z) różnią się w zależności od kontekstu komórkowego. Białko wiążące DNA, Pleiohomeotic (Pho), wraz z innymi czynnikami, wiąże się do elementów odpowiedzi Polycomb (PRE) genów docelowych, po czym E(z) trimetyluje histon H3K27. Chociaż PRE zostały zidentyfikowane tylko u Drosophila, metaanaliza przypuszczalnych genów docelowych dla białek PcG wykazała, że wiele genów docelowych jest wspólnych dla muchy, myszy i człowieka. dac i Egfr należą do tych genów (Schuettengruber i in., 2007). Tak więc region regulatorowy genu dac świerszcza prawdopodobnie zawiera PREs, poprzez które E(z) epigenetycznie reguluje ekspresję dac podczas regeneracji nóg świerszcza (Rysunek 2). Trwające badania koncentrują się na scharakteryzowaniu funkcji genu Pho i innych genów kompleksu PcG oraz modyfikatorów epigenetycznych podczas regeneracji nóg Gryllus.
Wreszcie, dlaczego E(z) RNAi powoduje powstawanie extra-tibia? Jednym z hipotetycznych scenariuszy jest to, że gdy piszczel jest amputowana w pozycji proksymalnej, gdzie ekspresja dac jest niska, ekspresja Utx (która dominuje w ekspresji E(z)) pozwala na ekspresję dac (rysunek 3a) w celu odtworzenia piszczeli. 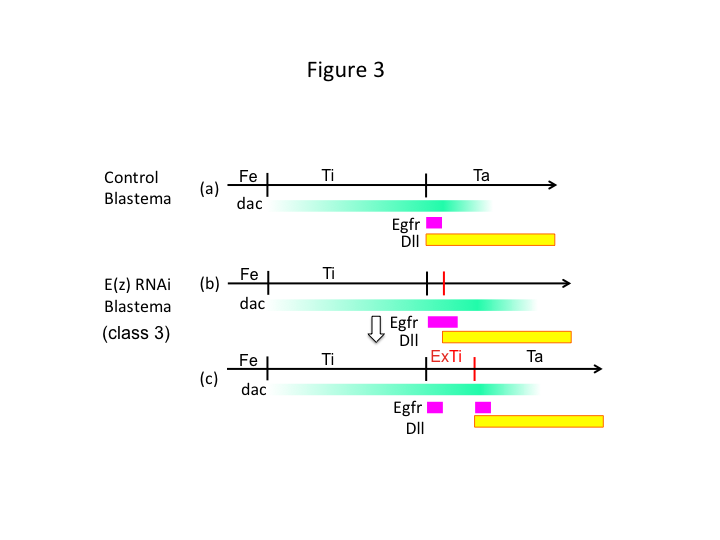 Tak więc, te modyfikatory histonowe wyczuwają wartości pozycji wzdłuż osi PD miejsca amputacji i dostrajają poziom ekspresji genów kształtujących nogi, jak dac. W przypadku E(z) RNAi tuż przed proksymalną amputacją, intensywna ekspresja dac jest indukowana i rozszerza się w regenerującej się nodze (Figura 3b). Ekspresja Distal-less (Dll), który jest innym genem luki w nodze, który określa dystalną domenę nogi (Angelini i Kaufman, 2005), może przesunąć się bardziej dystalnie w zależności od rozszerzonej ekspresji dac (Figura 3b). Tak więc domena Egfr może być podzielona na dwie części, gdzie (1) ekspresja Dll jest niska i (2) Dll jest wysoka. Extra-tibia prawdopodobnie tworzy się między dwoma różnymi Egfr-expressing domen przez intercalating proliferacji komórek i patterning (rysunek 3c).
Tak więc, te modyfikatory histonowe wyczuwają wartości pozycji wzdłuż osi PD miejsca amputacji i dostrajają poziom ekspresji genów kształtujących nogi, jak dac. W przypadku E(z) RNAi tuż przed proksymalną amputacją, intensywna ekspresja dac jest indukowana i rozszerza się w regenerującej się nodze (Figura 3b). Ekspresja Distal-less (Dll), który jest innym genem luki w nodze, który określa dystalną domenę nogi (Angelini i Kaufman, 2005), może przesunąć się bardziej dystalnie w zależności od rozszerzonej ekspresji dac (Figura 3b). Tak więc domena Egfr może być podzielona na dwie części, gdzie (1) ekspresja Dll jest niska i (2) Dll jest wysoka. Extra-tibia prawdopodobnie tworzy się między dwoma różnymi Egfr-expressing domen przez intercalating proliferacji komórek i patterning (rysunek 3c).
Naszym celem jest wyjaśnienie blueprints dla „podejmowania zregenerowaną nogę” przy użyciu tego atrakcyjnego modelu hemimetabolous owadów. Plany są oczekiwane, aby wyjaśnić, jak liczba segmentów nogi jest określona. Nasze uderzające obserwacje na temat RNAi przeciwko E(z) prowadzące do „tworzenia dodatkowych piszczeli” stanowią ważny krok w kierunku wyjaśnienia tego procesu.
- Mito, T. i Noji, S. (2008). The Two-Spotted Cricket Gryllus bimaculatus: An emerging Model for Developmental and Regeneration Studies. Cold Spring Harb Protoc, 331-346.
- Umesono, Y., Tasaki, J., Nishimura, Y., Hrouda, M., Kawaguchi, E., Yazawa, S., Nishimura, O., Hosoda, K., Inoue, T. and Agata, K. (2013). The molecular logic for planarian regeneration along the anterior-posterior axis. Nature 500, 73-76.
- Endo, T., Bryant, S. V. and Gardiner, D. M. (2004). A stepwise model system for limb regeneration. Dev Biol 270, 135-145.
- French, V., Bryant, P. J. and Bryant, S. V. (1976). Regulacja wzorów w polach epimorficznych. Science 193, 969-981.
- Bando, T., Ishimaru, Y., Kida, T., Hamada, Y., Matsuoka, Y., Nakamura, T., Ohuchi, H., Noji, S. and Mito, T. (2013). Analysis of RNA-Seq data reveals involvement of JAK/STAT signalling during leg regeneration in the cricket Gryllus bimaculatus. Development 140, 959-964.
- Hamada, Y., Bando, T., Nakamura, T., Ishimaru, Y., Mito, T., Noji, S., Tomioka, K. and Ohuchi, H. (2015). Leg regeneration is epigenetically regulated by histone H3K27 methylation in the cricket Gryllus bimaculatus. Development 142, 2916-2927.
- Ishimaru, Y., Nakamura, T., Bando, T., Matsuoka, Y., Ohuchi, H., Noji, S. and Mito, T. (2015). Involvement of dachshund and Distal-less in distal pattern formation of the cricket leg during regeneration. Sci Rep 5, 8387.
- Schuettengruber, B., Chourrout, D., Vervoort, M., Leblanc, B. and Cavalli, G. (2007). Genome regulation by polycomb and trithorax proteins. Cell 128, 735-745.
- Matsuoka, Y., Bando, T., Watanabe, T., Ishimaru, Y., Noji, S., Popadić, A. and Mito, T. (2015). Short germ insects utilize both the ancestral and derived mode of Polycomb group-mediated epigenetic silencing of Hox genes. Biol Open 4, 702-709.
- Angelini,R. and Kaufman, T. C. (2005). Insect appendages and comparative ontogenetics. Dev Biol 286, 57-77.
Share
- Issue:*
- Your Name:*
- Your Email:*
- Details:*
Submit Report
 (1 votes)
(1 votes)
Tags: Chromatyna i epigenetyka, świerszcz, regeneracja
Kategorie: Badania
.