Im Herbst zirpen die Grillen im gemäßigten ostasiatischen Land Japan. Die Afrikanische Feldgrille Gryllus bimaculatus stammt zwar aus tropischen Ländern, ist aber aufgrund ihrer Fähigkeit, amputierte Beine während der Nymphe zu regenerieren, und ihrer Entwicklungsweise (kurzes Keimband) weltweit ein aufstrebendes Modelltier (Mito und Noji, 2008).
Viele Lebewesen im Tierreich sind in der Lage, ihre Körperteile nach Verletzungen nachwachsen zu lassen. Beispiele für Körperteile, die nachwachsen können, sind die Linse und der Schwanz von Amphibien, der Kopf von Planarien und das Herz von Fischen. Im Gegensatz dazu wird seit langem davon ausgegangen, dass der Mensch verlorene Körperteile nicht wiederherstellen kann, mit Ausnahme bestimmter Gewebe wie der Epidermis, der Leber und der Eierstockoberfläche nach dem Eisprung. Daher ist es wichtig, die molekularen Mechanismen, die an Regenerationsprozessen beteiligt sind, anhand von Tiermodellen aufzuklären, die in der Lage sind, Körperteile zu regenerieren, um sie anschließend auf nicht regenerierbare menschliche Organe und Gewebe anzuwenden.
In den letzten zwei Jahren führten vergleichende Genomstudien an zwei Planarienarten mit unterschiedlichen Regenerationsfähigkeiten zur erfolgreichen Regeneration von Köpfen durch Verringerung der Beta-Catenin-Aktivität von ansonsten nicht regenerierbaren Schwanzfragmenten (Umesono et al., 2013). Studien an Wirbeltieren mit der Fähigkeit zur Wiederherstellung von Gliedmaßen, darunter Molche, Frösche und Salamander, haben gezeigt, dass die Gliedmaßenregeneration schrittweise erfolgt. Der Regenerationsprozess der Gliedmaßen wird in mindestens drei Phasen unterteilt: Wundheilung, Entdifferenzierung und Neuentwicklung, wobei die Neuentwicklungsphase die Embryonalentwicklung nachahmt (Endo et al., 2004).
Das Bein der Grille besteht aus sechs Segmenten, die entlang der proximal-distalen Achse (PD) angeordnet sind: Coxa, Trochanter, Femur, Tibia, Tarsus und Klaue (Abbildung 1). 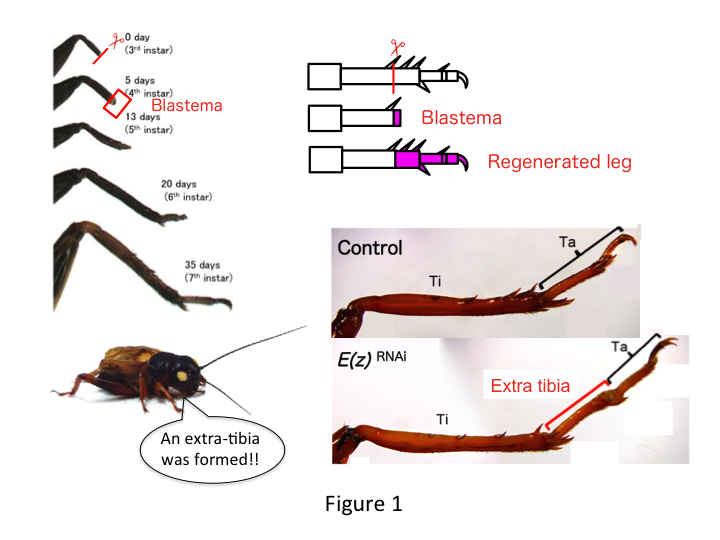 Der Tarsus ist weiter in drei Tarsomere unterteilt. Wird das Schienbein der Nymphe im dritten Larvenstadium amputiert, so regeneriert sich das Bein und nimmt bis zum sechsten Instadium (d. h. innerhalb von 20 Tagen nach der Amputation) seine allometrische Größe und Form wieder an, so dass es fast die normale Größe und Form eines erwachsenen Tieres erreicht. Bald nach der Heilung bildet sich im distalen Bereich des amputierten Beins ein Blastema (ein Pool von Zellen, die sich vermehren). Blastema-Zellen vermehren sich und bilden die fehlenden Strukturen durch interkalare Prozesse zwischen der distalsten Region und dem verbleibenden Teil des Beins (French et al., 1976).
Der Tarsus ist weiter in drei Tarsomere unterteilt. Wird das Schienbein der Nymphe im dritten Larvenstadium amputiert, so regeneriert sich das Bein und nimmt bis zum sechsten Instadium (d. h. innerhalb von 20 Tagen nach der Amputation) seine allometrische Größe und Form wieder an, so dass es fast die normale Größe und Form eines erwachsenen Tieres erreicht. Bald nach der Heilung bildet sich im distalen Bereich des amputierten Beins ein Blastema (ein Pool von Zellen, die sich vermehren). Blastema-Zellen vermehren sich und bilden die fehlenden Strukturen durch interkalare Prozesse zwischen der distalsten Region und dem verbleibenden Teil des Beins (French et al., 1976).
Zuvor haben wir eine vergleichende Transkriptomanalyse von regenerierenden und normalen amputierten Beinen von Grillen durchgeführt, um ein Profil der mRNA-Expression zu erstellen, die mit der Beinregeneration verbunden ist (Bando et al., 2013). Wir konzentrierten uns zunächst auf die Hochregulierung von Genen des Jak/Stat-Signalwegs, die mit dem Immunsystem verbunden sind. Die RNA-Interferenz (RNAi) von Genen in diesem Signalweg hat die Regeneration der Beine gründlich gestört. Im Gegensatz dazu bewirkte RNAi gegen Socs, einen Unterdrücker der Zytokin-Signalübertragung, eine Verlängerung der Beine. Weitere Experimente zeigten, dass der Jak/Stat-Signalweg die Zellproliferation stromabwärts des Ds/Fat-Signalwegs fördert.
Anschließend untersuchten wir die epigenetische Regulierung während der Beinregeneration der Grille. Tetsuya Bando, ein leitender Forscher unserer Gruppe, identifizierte ein Gen für die Histon H3-Lysin 27 (H3K27) Methyltransferase, E(z), und ein Gen für die Histon H3K27 Demethylase, Utx, in G. bimaculatus. Das Klonen von Gryllus-Genen ist jetzt ein einfacher Prozess, da Informationen über das Genom der Grille verfügbar sind (Mito und Noji, persönliche Mitteilung). Die Methylierung des Histons H3K27 durch E(z) unterdrückt die Expression von Zielgenen durch die Rekrutierung von Proteinen der Polycomb-Gruppe. Umgekehrt fördert die Demethylierung des trimethylierten Histons H3K27 durch Utx die Genexpression. Tetsuya fand heraus, dass die Transkription sowohl der E(z)- als auch der Utx-Gene in den Blastema-Zellen amputierter Beine hochreguliert ist (Bando et al., 2013). Durch In-situ-Hybridisierung wurde nachgewiesen, dass beide Gene in den regenerierenden Beinen von Grillen ubiquitär transkribiert werden und dass beide Gene in sich entwickelnden Embryonen exprimiert werden (Hamada et al., 2015). Immunfärbungen an den amputierten Beinchen nach RNAi durch Yoshimasa Hamada (ein Doktorand) bestätigten, dass E(z) und Utx zur Methylierung bzw. Demethylierung am Histon H3K27me3 während der Beinregeneration beitragen.
Allerdings stellte Yoshimasa unerwartet fest, dass das zusätzliche Beinsegment nach RNAi gegen E(z) gebildet wird (Abbildung 1). Zunächst war es uns nicht möglich, die Identität des Beinsegments zu bestimmen. Morphologisch schien das Beinsegment ein Schienbein zu sein, da es Stacheln und Sporne hatte, die für ein echtes Schienbein charakteristisch sind. Unsere erste Hypothese war, dass die Phänotypen nach RNAi von der Amputationsstelle im Schienbein abhängen könnten. Doch selbst wenn ein Bein im distalen Teil des Oberschenkels amputiert wird, entsteht ein zusätzliches tibiaähnliches Segment. Die Musterbildung entlang der antero-posterioren und dorso-ventralen Achsen blieb unverändert, außer entlang der PD-Achse. Anschließend untersuchten wir, ob die Amputationsstelle entlang der PD-Achse in der Tibia den Schweregrad des Phänotyps beeinflusst. Die sich bildende Extra-Tibia wurde umso länger, je proximaler die Amputationsstellen an der Tibia waren (Abbildung 1). Umgekehrt führte RNAi gegen Utx zu einem Verlust der Gelenkbildung zwischen Tarsomere 1 (Ta1) und Ta2 (Abbildung 2). 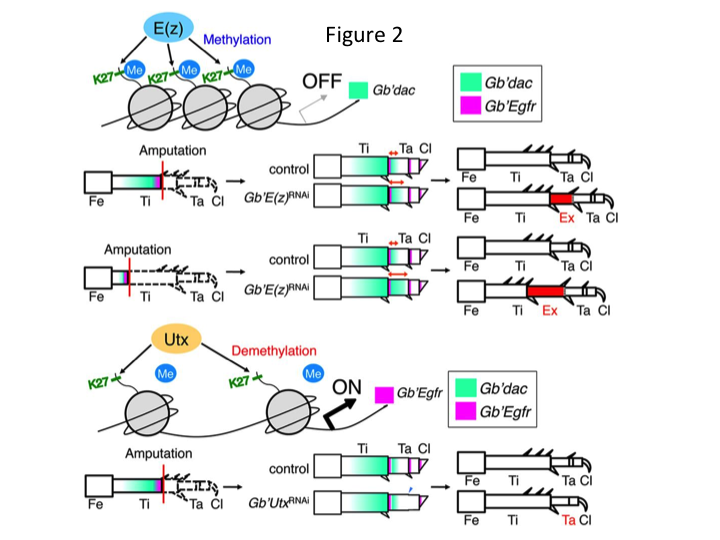 Die In-situ-Hybridisierung zeigte, dass sich die Expression von Genen für die Beinformung entlang der PD-Achse veränderte. Insbesondere nahm die Expression von Dac in den sich regenerierenden E(z)RNAi-Beinen zu, während die Expression von Egfr in UtxRNAi-Beinen abnahm. Daher kann E(z) die dac-Expression während der normalen Beinregeneration unterdrücken, während Utx die Egfr-Expression induziert.
Die In-situ-Hybridisierung zeigte, dass sich die Expression von Genen für die Beinformung entlang der PD-Achse veränderte. Insbesondere nahm die Expression von Dac in den sich regenerierenden E(z)RNAi-Beinen zu, während die Expression von Egfr in UtxRNAi-Beinen abnahm. Daher kann E(z) die dac-Expression während der normalen Beinregeneration unterdrücken, während Utx die Egfr-Expression induziert.
dac kodiert für einen transkriptionellen Co-Repressor, der in Beinlückengenen kategorisiert ist. dac erzeugt grobe Positionswerte entlang der PD-Achse des Beins und vermittelt die Bildung der distalen Tibia und Ta1 (das proximale Tarsomer) während der Regeneration des Grillenbeins (dac-Expressionsdomäne ist in Abbildung 2 grün dargestellt) (Ishimaru et al., 2015). Insbesondere fördert dac die Proliferation von Tibiazellen. Da RNAi gegen E(z) dac hochreguliert, könnte die E(z)-Expression in den Blastemazellen die blastemale Überproliferation unterdrücken, indem sie die zusätzliche dac-Expression unterdrückt.
Diese Information wirft die Frage auf, wie E(z) spezifisch die dac-Expression reguliert. Außerdem stellt sich die Frage, welcher Mechanismus die Zielgene von E(z) bestimmt. E(z) gehört zum Polycomb repressiven Komplex 2 (PRC2), einem der drei Komplexe der Polycomb-Gruppe (PcG) (Schuettengruber et al., 2007). Während der Cricket-Embryogenese unterdrückt E(z) die anteriore Expansion der Hox-Genexpression und sorgt für die richtige Identität der Embryonen (Matsuoka et al., 2015). Diese Informationen deuten darauf hin, dass sich die Zielgene von E(z) je nach zellulärem Kontext unterscheiden. Ein DNA-bindendes Protein, Pleiohomeotic (Pho), bindet zusammen mit anderen Faktoren an die Polycomb-Response-Elemente (PRE) der Zielgene, woraufhin E(z) das Histon H3K27 trimethyliert. Obwohl PREs nur in Drosophila identifiziert wurden, hat die Meta-Analyse von mutmaßlichen Zielgenen für PcG-Proteine gezeigt, dass viele der Zielgene in Fliege, Maus und Mensch vorkommen. dac und Egfr gehören zu diesen Genen (Schuettengruber et al., 2007). Somit enthält die regulatorische Region des dac-Gens der Grille wahrscheinlich PREs, durch die E(z) die Expression von dac während der Regeneration der Grillenbeine epigenetisch reguliert (Abbildung 2). Laufende Forschungsarbeiten konzentrieren sich auf die Charakterisierung der Funktionen des Pho-Gens und anderer Gene des PcG-Komplexes sowie epigenetischer Modifikatoren während der Regeneration der Gryllus-Beine.
Warum verursacht E(z) RNAi schließlich die Bildung von Extra-Tibien? Ein hypothetisches Szenario ist, dass, wenn die Tibia an der proximalen Position amputiert wird, wo die dac-Expression niedrig ist, die Utx-Expression (die die E(z)-Expression dominiert) die dac-Expression ermöglicht (Abbildung 3a), um die Tibia wiederherzustellen. 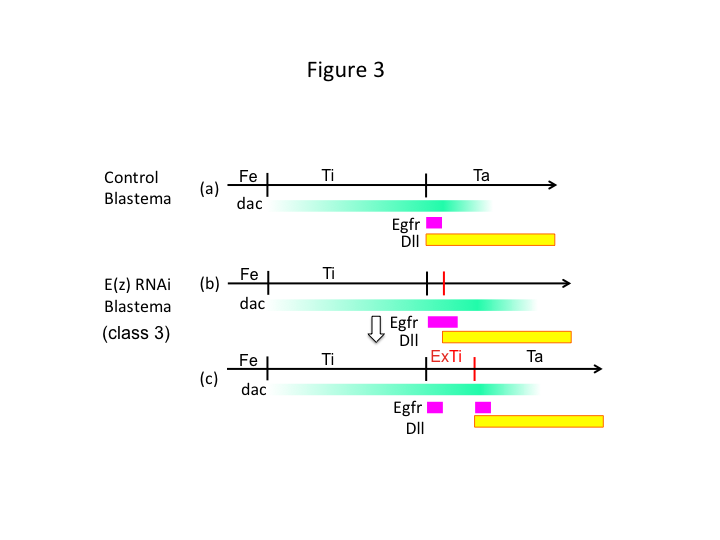 Diese Histon-Modifikatoren erkennen also die Positionswerte entlang der PD-Achse der Amputationsstelle und nehmen eine Feinabstimmung des Expressionsniveaus von Genen für die Beinformung, wie dac, vor. Im Falle von E(z)-RNAi kurz vor der proximalen Amputation wird eine intensive dac-Expression induziert, die sich im regenerierenden Bein ausbreitet (Abbildung 3b). Die Expression von Distal-less (Dll), einem weiteren Beinlückengen, das den distalen Bereich des Beins spezifiziert (Angelini und Kaufman, 2005), kann sich in Abhängigkeit von der erweiterten dac-Expression weiter nach distal verlagern (Abbildung 3b). Somit kann der Egfr-exprimierende Bereich in zwei Teile aufgeteilt werden, in denen (1) die Dll-Expression niedrig und (2) die Dll-Expression hoch ist. Die Extra-Tibia bildet sich wahrscheinlich zwischen den beiden unterschiedlichen Egfr-exprimierenden Domänen durch interkalierende Zellproliferation und Musterung (Abbildung 3c).
Diese Histon-Modifikatoren erkennen also die Positionswerte entlang der PD-Achse der Amputationsstelle und nehmen eine Feinabstimmung des Expressionsniveaus von Genen für die Beinformung, wie dac, vor. Im Falle von E(z)-RNAi kurz vor der proximalen Amputation wird eine intensive dac-Expression induziert, die sich im regenerierenden Bein ausbreitet (Abbildung 3b). Die Expression von Distal-less (Dll), einem weiteren Beinlückengen, das den distalen Bereich des Beins spezifiziert (Angelini und Kaufman, 2005), kann sich in Abhängigkeit von der erweiterten dac-Expression weiter nach distal verlagern (Abbildung 3b). Somit kann der Egfr-exprimierende Bereich in zwei Teile aufgeteilt werden, in denen (1) die Dll-Expression niedrig und (2) die Dll-Expression hoch ist. Die Extra-Tibia bildet sich wahrscheinlich zwischen den beiden unterschiedlichen Egfr-exprimierenden Domänen durch interkalierende Zellproliferation und Musterung (Abbildung 3c).
Unser Ziel ist es, mit Hilfe dieses attraktiven hemimetabolischen Insektenmodells die Baupläne für die „Herstellung eines regenerierten Beins“ zu erforschen. Die Baupläne sollen klären, wie die Anzahl der Beinsegmente bestimmt wird. Unsere auffälligen Beobachtungen, dass RNAi gegen E(z) zur „Bildung zusätzlicher Schienbeine“ führt, sind ein wichtiger Schritt zur Aufklärung dieses Prozesses.
- Mito, T. and Noji, S. (2008). The Two-spotted Cricket Gryllus bimaculatus: An emerging Model for Developmental and Regeneration Studies. Cold Spring Harb Protoc, 331-346.
- Umesono, Y., Tasaki, J., Nishimura, Y., Hrouda, M., Kawaguchi, E., Yazawa, S., Nishimura, O., Hosoda, K., Inoue, T. und Agata, K. (2013). Die molekulare Logik für die Regeneration von Planarien entlang der anterior-posterioren Achse. Nature 500, 73-76.
- Endo, T., Bryant, S. V. und Gardiner, D. M. (2004). Ein schrittweises Modellsystem für die Regeneration von Gliedmaßen. Dev Biol 270, 135-145.
- French, V., Bryant, P. J. und Bryant, S. V. (1976). Musterregulation in epimorphen Feldern. Science 193, 969-981.
- Bando, T., Ishimaru, Y., Kida, T., Hamada, Y., Matsuoka, Y., Nakamura, T., Ohuchi, H., Noji, S. und Mito, T. (2013). Die Analyse von RNA-Seq-Daten zeigt die Beteiligung von JAK/STAT-Signalen während der Beinregeneration bei der Grille Gryllus bimaculatus. Development 140, 959-964.
- Hamada, Y., Bando, T., Nakamura, T., Ishimaru, Y., Mito, T., Noji, S., Tomioka, K. und Ohuchi, H. (2015). Beinregeneration wird epigenetisch durch Histon H3K27-Methylierung bei der Grille Gryllus bimaculatus reguliert. Development 142, 2916-2927.
- Ishimaru, Y., Nakamura, T., Bando, T., Matsuoka, Y., Ohuchi, H., Noji, S. und Mito, T. (2015). Die Beteiligung von Dackel und Distal-less an der distalen Musterbildung des Grillenbeins während der Regeneration. Sci Rep 5, 8387.
- Schuettengruber, B., Chourrout, D., Vervoort, M., Leblanc, B. and Cavalli, G. (2007). Genomregulation durch Polycomb- und Trithorax-Proteine. Cell 128, 735-745.
- Matsuoka, Y., Bando, T., Watanabe, T., Ishimaru, Y., Noji, S., Popadić, A. and Mito, T. (2015). Short germ insects utilize both the ancestral and derived mode of Polycomb group-mediated epigenetic silencing of Hox genes. Biol Open 4, 702-709.
- Angelini,R. and Kaufman, T. C. (2005). Insect appendages and comparative ontogenetics. Dev Biol 286, 57-77.
Teilen
- Ausgabe:*
- Ihr Name:*
- Ihre E-Mail:*
- Details:*
Bericht einreichen
 (1 votes)
(1 votes)
Tags: Chromatin und Epigenetik, Grille, Regeneration
Kategorien: Forschung