In autunno, i grilli generalmente esibiscono canti fruscianti nel paese temperato dell’Asia orientale del Giappone. Mentre il grillo africano Gryllus bimaculatus è originario dei paesi tropicali, è un animale modello emergente a livello globale a causa della sua capacità di rigenerare le gambe amputate durante la ninfa e la sua modalità di sviluppo (banda germinale corta) (Mito e Noji, 2008).
Molti organismi viventi nel regno animale sono in grado di far ricrescere le loro parti del corpo dopo una lesione. Esempi di parti del corpo che possono essere ricresciute sono il cristallino e la coda degli anfibi, la testa dei planari e il cuore dei pesci. Al contrario, è stato a lungo ipotizzato che gli esseri umani non possono ripristinare le parti del corpo perse, ad eccezione di particolari tessuti, tra cui l’epidermide, il fegato e la superficie ovarica dopo l’ovulazione. Pertanto, è importante chiarire i meccanismi molecolari coinvolti nei processi di rigenerazione utilizzando modelli animali che sono in grado di rigenerare parti del corpo per la successiva applicazione in organi e tessuti umani non rigenerabili.
Negli ultimi 2 anni, studi genomici comparativi di due specie di planarie con diverse capacità rigenerative hanno portato alla rigenerazione con successo di teste riducendo l’attività della beta-catenina da frammenti di coda altrimenti non rigenerabili (Umesono et al., 2013). Studi di vertebrati con la capacità di ripristinare gli arti, tra cui tritoni, rane e salamandre, hanno dimostrato che la rigenerazione degli arti avviene in modo graduale. Il processo di rigenerazione degli arti è diviso in almeno tre fasi: guarigione della ferita, de-differenziazione e ri-sviluppo, con la fase di ri-sviluppo che imita lo sviluppo embrionale (Endo et al., 2004).
La gamba del grillo è composta da sei segmenti che sono disposti lungo l’asse prossimale-distale (PD): coxa, trocantere, femore, tibia, tarso e artiglio (Figura 1). 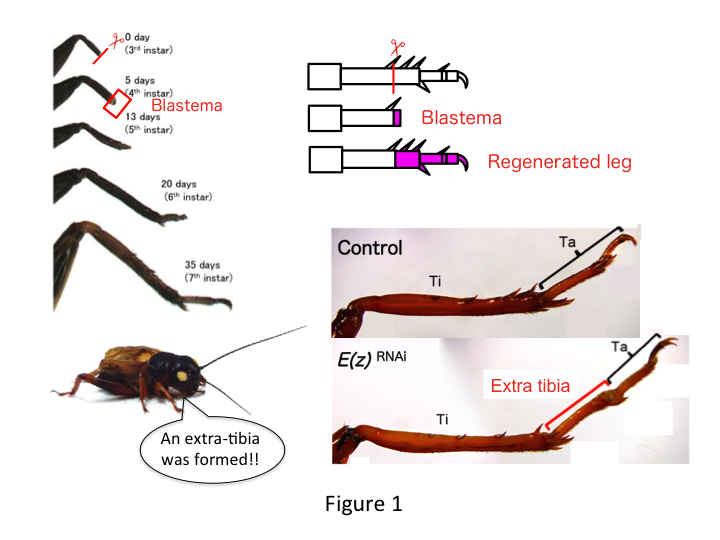 Il tarso è ulteriormente suddiviso in tre tarsomeri. Quando la tibia della ninfa al terzo stadio viene amputata, la gamba si rigenera e recupera le sue dimensioni allometriche e la forma corretta entro il sesto stadio (cioè entro 20 giorni dall’amputazione), venendo ripristinata a dimensioni e forma quasi normali per gli adulti. Subito dopo la guarigione, il blastema (un pool di cellule che proliferano) si sviluppa nella regione distale della gamba amputata. Le cellule del blastema proliferano e formano le strutture mancanti tramite processi intercalari tra la regione più distale e la parte rimanente della gamba (French et al., 1976).
Il tarso è ulteriormente suddiviso in tre tarsomeri. Quando la tibia della ninfa al terzo stadio viene amputata, la gamba si rigenera e recupera le sue dimensioni allometriche e la forma corretta entro il sesto stadio (cioè entro 20 giorni dall’amputazione), venendo ripristinata a dimensioni e forma quasi normali per gli adulti. Subito dopo la guarigione, il blastema (un pool di cellule che proliferano) si sviluppa nella regione distale della gamba amputata. Le cellule del blastema proliferano e formano le strutture mancanti tramite processi intercalari tra la regione più distale e la parte rimanente della gamba (French et al., 1976).
In precedenza, abbiamo eseguito un’analisi comparativa del trascrittoma delle zampe amputate normali e rigeneranti dei grilli per tracciare il profilo dell’espressione dell’mRNA associato alla rigenerazione della gamba (Bando et al., 2013). Per prima cosa ci siamo concentrati sull’upregolazione dei geni della via Jak/Stat, che sono legati al sistema immunitario. RNA interference (RNAi) dei geni in questo percorso completamente disturbato rigenerazione gamba. Al contrario, l’RNAi contro Socs, un soppressore della segnalazione delle citochine, ha causato l’allungamento delle gambe. Ulteriori esperimenti hanno dimostrato che la via Jak/Stat promuove la proliferazione cellulare a valle della via Ds/Fat.
Di conseguenza, abbiamo studiato la regolazione epigenetica durante la rigenerazione delle gambe del grillo. Tetsuya Bando, un ricercatore senior del nostro gruppo, ha identificato un gene per l’istone H3 lisina 27 (H3K27) metiltransferasi, E(z), e un gene per l’istone H3K27 demetilasi, Utx, in G. bimaculatus. Clonare i geni di Gryllus è ora un processo semplice grazie alle informazioni disponibili sul genoma del grillo (Mito e Noji, comunicazione personale). La metilazione dell’istone H3K27 da parte di E(z) reprime l’espressione dei geni bersaglio reclutando le proteine del gruppo Polycomb. Al contrario, la demetilazione dell’istone trimetilato H3K27 da parte di Utx promuove l’espressione dei geni. Tetsuya ha scoperto che la trascrizione di entrambi i geni E(z) e Utx è upregolata nelle cellule del blastema delle gambe amputate (Bando et al., 2013). L’ibridazione in situ ha verificato che entrambi i geni sono trascritti ubiquitariamente nelle gambe rigeneranti dei grilli, e che entrambi i geni sono espressi negli embrioni in via di sviluppo (Hamada et al., 2015). L’immunostaining sulle zampe minuscole amputate dopo RNAi da Yoshimasa Hamada (uno studente di dottorato) ha confermato che E(z) e Utx contribuiscono alla metilazione e demetilazione all’istone H3K27me3, rispettivamente, durante la rigenerazione delle gambe.
Tuttavia, Yoshimasa ha inaspettatamente trovato che il segmento di gamba extra si forma dopo RNAi contro E(z) (Figura 1). Inizialmente, non siamo stati in grado di determinare l’identità del segmento della gamba. Morfologicamente, il segmento della gamba sembrava essere una tibia, perché aveva spine e speroni che erano caratteristici di una tibia autentica. La nostra ipotesi iniziale era che i fenotipi dopo RNAi potessero dipendere dal sito di amputazione nella tibia. Tuttavia, anche quando una gamba viene amputata nella parte distale del femore, il segmento extra simile alla tibia emerge. La formazione del modello lungo gli assi antero-posteriore e dorso-ventrale è rimasta invariata, tranne che lungo l’asse PD. Abbiamo poi esaminato se il sito di amputazione lungo l’asse PD nella tibia ha influenzato la gravità fenotipica. L’extra-tibia che si formava diventava più lunga quanto più prossimali erano i siti di amputazione sulla tibia (Figura 1). Al contrario, l’RNAi contro Utx ha portato alla perdita della formazione dell’articolazione tra il tarsomero 1 (Ta1) e Ta2 (Figura 2). 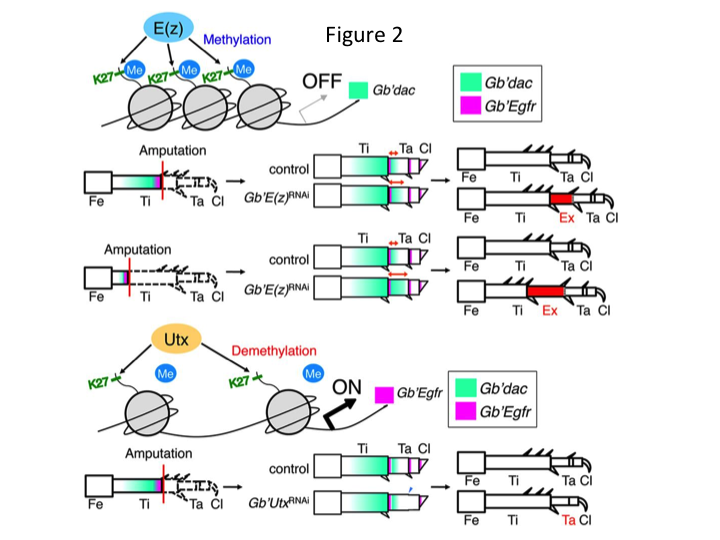 L’ibridazione in situ ha mostrato che l’espressione dei geni del patterning delle gambe si alterava lungo l’asse PD. In particolare, il dominio di espressione bassotto (dac) espanso in E (z) RNAi rigenerazione gambe, mentre l’espressione Egfr diminuito in UtxRNAi gambe. Pertanto, E (z) può reprimere l’espressione dac durante la rigenerazione della gamba normale, mentre Utx induce l’espressione Egfr.
L’ibridazione in situ ha mostrato che l’espressione dei geni del patterning delle gambe si alterava lungo l’asse PD. In particolare, il dominio di espressione bassotto (dac) espanso in E (z) RNAi rigenerazione gambe, mentre l’espressione Egfr diminuito in UtxRNAi gambe. Pertanto, E (z) può reprimere l’espressione dac durante la rigenerazione della gamba normale, mentre Utx induce l’espressione Egfr.
dac codifica un co-repressore trascrizionale che è classificato nei geni di gap gamba. dac produce valori posizionali grezzi lungo l’asse PD della gamba e media la formazione della tibia distale e Ta1 (il tarsomero prossimale) durante la rigenerazione gamba grillo (dominio di espressione dac è mostrato in verde nella Figura 2) (Ishimaru et al., 2015). In particolare, dac promuove la proliferazione delle cellule tibiali. Pertanto, poiché RNAi contro E(z) upregulates dac, l’espressione di E(z) nelle cellule del blastema può sopprimere la sovrapproliferazione blastemica reprimendo l’espressione extra dac.
Queste informazioni sollevano la questione di come E(z) regola specificamente l’espressione dac. Inoltre, qual è il meccanismo che determina i geni bersaglio di E(z)? E(z) appartiene al complesso repressivo Polycomb 2 (PRC2), che è uno dei tre complessi del gruppo Polycomb (PcG) (Schuettengruber et al., 2007). Durante l’embriogenesi del grillo, E(z) reprime l’espansione anteriore dell’espressione del gene Hox e fornisce una corretta identità negli embrioni (Matsuoka et al., 2015). Queste informazioni indicano che i geni bersaglio di E(z) differiscono a seconda del contesto cellulare. Una proteina legante il DNA, Pleiohomeotic (Pho), insieme ad altri fattori, si lega agli elementi di risposta Polycomb (PRE) dei geni bersaglio, dopo di che E(z) trimetila l’istone H3K27. Anche se i PRE sono stati identificati solo in Drosophila, la meta-analisi dei geni bersaglio putativi per le proteine PcG ha dimostrato che molti dei geni bersaglio sono comuni alla mosca, al topo e all’uomo. dac e Egfr sono inclusi tra questi geni (Schuettengruber et al., 2007). Così, la regione di regolazione del gene dac grillo contiene probabilmente PREs, attraverso il quale E (z) regola epigeneticamente l’espressione di dac durante la rigenerazione gamba grillo (Figura 2). La ricerca in corso si concentra sulla caratterizzazione delle funzioni del gene Pho e di altri geni del complesso PcG e dei modificatori epigenetici durante la rigenerazione delle zampe di Gryllus.
Infine, perché E(z) RNAi causa la formazione di extra-tibia? Uno scenario ipotetico è che quando la tibia viene amputata nella posizione prossimale dove l’espressione di dac è bassa, l’espressione di Utx (che domina l’espressione di E(z)) permette l’espressione di dac (Figura 3a) per ripristinare la tibia. 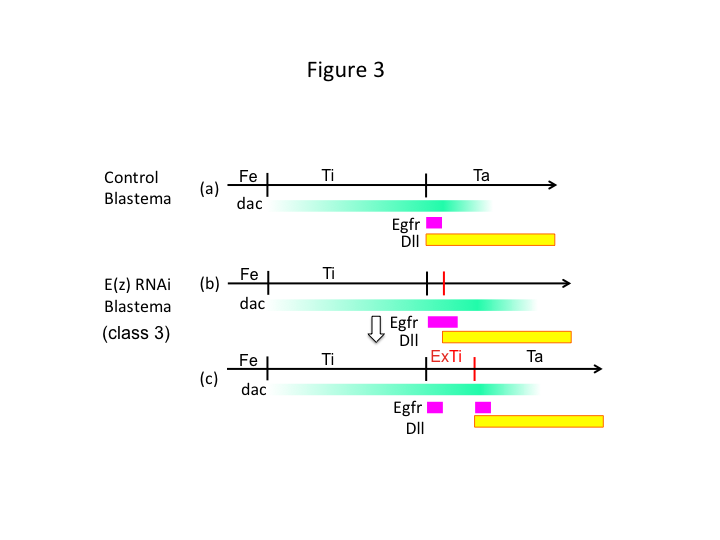 Quindi, questi modificatori istonici percepiscono i valori posizionali lungo l’asse PD del sito di amputazione, e regolano il livello di espressione dei geni del patterning delle gambe, come dac. Nel caso di E(z) RNAi appena prima dell’amputazione prossimale, l’intensa espressione di dac è indotta e si espande nella gamba rigenerante (Figura 3b). Distal-less (Dll) espressione, che è un altro gene gap gamba che specifica il dominio distale della gamba (Angelini e Kaufman, 2005), può spostare più distalmente a seconda dell’espressione dac espansa (Figura 3b). Così, il dominio che esprime Egfr può essere separato in due parti dove (1) l’espressione di Dll è bassa e (2) Dll è alta. L’extra-tibia si forma probabilmente tra i due diversi domini che esprimono Egfr attraverso la proliferazione cellulare intercalare e il patterning (Figura 3c).
Quindi, questi modificatori istonici percepiscono i valori posizionali lungo l’asse PD del sito di amputazione, e regolano il livello di espressione dei geni del patterning delle gambe, come dac. Nel caso di E(z) RNAi appena prima dell’amputazione prossimale, l’intensa espressione di dac è indotta e si espande nella gamba rigenerante (Figura 3b). Distal-less (Dll) espressione, che è un altro gene gap gamba che specifica il dominio distale della gamba (Angelini e Kaufman, 2005), può spostare più distalmente a seconda dell’espressione dac espansa (Figura 3b). Così, il dominio che esprime Egfr può essere separato in due parti dove (1) l’espressione di Dll è bassa e (2) Dll è alta. L’extra-tibia si forma probabilmente tra i due diversi domini che esprimono Egfr attraverso la proliferazione cellulare intercalare e il patterning (Figura 3c).
Il nostro obiettivo è quello di chiarire i progetti per “fare una gamba rigenerata” utilizzando questo attraente modello di insetto emimetabolo. I progetti dovrebbero chiarire come viene determinato il numero di segmenti delle gambe. Le nostre osservazioni sorprendenti sull’RNAi contro E(z) che porta alla “formazione di tibia extra” rappresentano un passo importante verso la spiegazione di questo processo.
- Mito, T. e Noji, S. (2008). Il grillo a due macchie Gryllus bimaculatus: Un modello emergente per studi di sviluppo e rigenerazione. Cold Spring Harb Protoc, 331-346.
- Umesono, Y., Tasaki, J., Nishimura, Y., Hrouda, M., Kawaguchi, E., Yazawa, S., Nishimura, O., Hosoda, K., Inoue, T. e Agata, K. (2013). La logica molecolare per la rigenerazione planare lungo l’asse anteriore-posteriore. Nature 500, 73-76.
- Endo, T., Bryant, S. V. e Gardiner, D. M. (2004). Un sistema di modello graduale per la rigenerazione degli arti. Dev Biol 270, 135-145.
- French, V., Bryant, P. J. and Bryant, S. V. (1976). Regolazione dei modelli nei campi epimorfi. Scienza 193, 969-981.
- Bando, T., Ishimaru, Y., Kida, T., Hamada, Y., Matsuoka, Y., Nakamura, T., Ohuchi, H., Noji, S. e Mito, T. (2013). Analisi di RNA-Seq dati rivela il coinvolgimento della segnalazione JAK/STAT durante la rigenerazione delle gambe nel grillo Gryllus bimaculatus. Sviluppo 140, 959-964.
- Hamada, Y., Bando, T., Nakamura, T., Ishimaru, Y., Mito, T., Noji, S., Tomioka, K. e Ohuchi, H. (2015). La rigenerazione delle gambe è epigeneticamente regolata dalla metilazione dell’istone H3K27 nel grillo Gryllus bimaculatus. Sviluppo 142, 2916-2927.
- Ishimaru, Y., Nakamura, T., Bando, T., Matsuoka, Y., Ohuchi, H., Noji, S. e Mito, T. (2015). Coinvolgimento di bassotto e Distal-less nella formazione del modello distale della gamba del grillo durante la rigenerazione. Sci Rep 5, 8387.
- Schuettengruber, B., Chourrout, D., Vervoort, M., Leblanc, B. e Cavalli, G. (2007). Regolazione del genoma da proteine polycomb e trithorax. Cell 128, 735-745.
- Matsuoka, Y., Bando, T., Watanabe, T., Ishimaru, Y., Noji, S., Popadić, A. e Mito, T. (2015). Breve insetti germe utilizzare sia la modalità ancestrale e derivata di Polycomb gruppo-mediata silenziamento epigenetico dei geni Hox. Biol Open 4, 702-709.
- Angelini,R. e Kaufman, T. C. (2005). Appendici degli insetti e ontogenetica comparativa. Dev Biol 286, 57-77.
Condividi
- Argomento:*
- Il tuo nome:*
- La tua email:*
- Dettagli:*
Invia segnalazione
 (1 votes)
(1 votes)
Tags: Cromatina ed epigenetica, grillo, rigenerazione
Categorie: Ricerca