Manuskript accepterat :29 Aug 19
Publicerat online den: 18-09-2019 Plagiatskontroll: Ja
Reviewed by: Farooq Sher
Tillbaka till granskning av: Grigorios Kyriakopoulos
Finellt godkännande av: Ayush Dogra
How to Citeclose | Publication Historyclose
Vijitra Luang-In1* , Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 och Kedsukon Maneewan1
, Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 och Kedsukon Maneewan1
1Natural Antioxidant Innovation Research Unit, Department of Biotechnology, Faculty of Technology, Mahasarakham University, Khamriang, Kantarawichai, Maha Sarakham, 44150, Thailand.
2International College of Digital Innovation, Chiang Mai University, Chiang Mai 50200, Thailand.
Korresponderande författare E-post: [email protected]
DOI : https://dx.doi.org/10.13005/bpj/1735
Abstract
Denna studie syftade till att isolera och identifiera bakterier som kan producera amylasenzym från den outforskade Nasinuanskogen, Kantarawichai-distriktet, Mahasarakhamprovinsen, Thailand. Tretton bakterieisolat med amylasproducerande förmåga på 1 % stärkelseagar identifierades med hjälp av 16S rRNA-sekvensering. Tolv bakterier var grampositiva, stavformade och identifierades som Bacillus spp. och en bakterie med gramnegativ och stavformad karaktär var Enterobacter cloacae. Deras närmaste släktingar hittades i Indien, Kina, Korea, Indonesien, Argentina, Italien, Israel, USA, Argentina och Sydafrika. Dessa bakterier testades för specifik amylasaktivitet efter 1-3 dagars enzyminduktion med 1 % stärkelse vid 37 °C. Resultaten visade den högsta specifika aktiviteten vid inkubering dag 2 i följande ordning: Bacillus cereus 3.5AL2 > 3.4AL1 > 1.4AL3 och därför valdes 2-dagars enzyminduktion för vidare analys. Bacillus sp. 3.5AL2 visade sig uppvisa den högsta specifika amylasenzymaktiviteten på 1,97 ± 0,41 U/mg protein vid de optimala förhållandena 60 °C och pH 7,0 efter 30 minuters inkubation med 1 % stärkelse i 0,05 M PBS-buffert. Denna amylasproducerande bakteriestam erbjuder stor potential för tillämpningar inom livsmedels- och jordbruksindustrin i Thailand.
Nyckelord
Amylas; Bakterier; Nasinuanskog; jord
Ladda ner denna artikel som:![]()
Copy the following to cite this article:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3).
Kopiera följande för att citera denna URL:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3). Available from: http://biomedpharmajournal.org/?p=28497
Introduktion
De stärkelsehydrolytiska amylaserna (α-amylas, β-amylas och glukoamylas) är några av de mest använda enzymerna i dagens bioteknik. Glukoamylaser (1,4-α-D-glukan glukanohydrolas; EC 3.2.1.3) eller amyloglukosidaser, är exo-verkande amylaser som producerar glukos från den icke-reducerande änden av stärkelse och motsvarande oligosackarider. Glukoamylaser används kommersiellt för omvandling av malto-oligosackarider till glukos1. Även om amylaser produceras från olika källor (mikroorganismer, växter och djur) är amylaser från mikrober mest lämpade för industriell produktion på grund av mikrobiell kort tillväxtperiod, låg kostnadseffektiv produktion, miljövänligt beteende, mindre hanteringsproblem för arbetstagarna, produktivitet2 och enkel manipulering av bakteriegener3. Bakterier och svampar tenderar att utsöndra amylaser utanför cellerna för att utföra extracellulär nedbrytning av stärkelse till sockerarter. Ökad industriell efterfrågan på mikrobiella amylaser har observerats på grund av deras specifika reaktion, milda förutsättningar för reaktionen och mindre energiförbrukning än de konventionella icke-enzymatiska kemiska metoderna.
Den omfattande tillämpningen av amylas inom livsmedels-, stärkelseförvätsknings-, förkalknings-, bryggeri-, tvättmedels-, pappers-, textil- och destillationsindustrierna har lett till att man har uppmärksammat en ökad inhemsk produktion av α-amylas4. Bacillus är en vanlig bakteriekälla för industriell amylasproduktion. Olika stammar har dock olika optimala tillväxtförhållanden och enzymproduktionsprofil. Enligt uppgift har Bacillus-stammar i stor utsträckning använts industriellt för att producera α-amylas, däribland B. amyloliquefaciens, B. subtilis5, B. licheniformis6, B. stearothermophilus7, B. megaterium8 och B. circulans9.
Hittills har ett antal amylasproducerande bakterier dokumenterats, men ingen studie om amylasproducerande bakterier från jord i Nasinuan Community Forest, Kantharawichai District, Mahasarakham Province, Thailand har rapporterats. Denna skog verkar vara rik på mikrobiell biologisk mångfald som kan vara användbar för produktion av industriella enzymer, inklusive amylas. Detta är den första rapporten om identifiering av amylasproducerande bakterier som isolerats från Nasinuan Forest. Dessa bakteriella amylaser kan i framtiden användas lokalt inom olika industrier som massa och papper, textil, bioetanol, bryggeri, livsmedelsbearbetning, djurfoder och jordbruk i Thailand.
Material och metoder
Jordprover
Jordprover samlades slumpmässigt in under markytan 15 cm och förvarades i polystyrenpåsar från Nasinuan Community Forest, Kantarawichai District, Mahasarakham Province, Thailand (yta på 9.6 hektar; koordinat 16.340941, 103.210799).
Isolering av amylasproducerande bakterier
Jordprovet (10 g) suspenderades i 90 mL steril 0,85 % NaCl-lösning. Suspensionerna (100 µL) av seriella spädningar spreds på stärkelseagar (g/L); 3,0 nötköttsextrakt, 10,0 löslig stärkelse, 15,0 agar pH 7,0 och inkuberades vid 37 ºC i 7 dagar. Därefter översvämmades plattorna med Grams jod. Alla kolonier där det bildades en klar zon runt kolonierna subkulturerades i flytande buljong och ströks minst fem gånger för att erhålla rena isolat, vilket bekräftades genom gramfärgning och 1000x ljusmikroskopisk observation. De rena isolaten inokulerades på stärkelseagar och inkuberades vid 37 °C i 7 dagar. Diametern på de tydliga zonerna över diametern på kolonierna mättes med hjälp av en linjal som Halo : Koloniförhållandet.
16S rRNA-gensekvensering och fylogenetisk analys
Rena bakterieisolat identifierades med hjälp av genomiskt DNA som erhållits genom ovanstående metod och universella primers: forward primer 27F 5′-GAGAGAGTTTGATYCTGGCTCAG-3′ och reverse primer 1492R 5’AAGGAGGTGATCCARCCGCA -3′. I 25 µl PCR-blandning bestod den av genomiskt DNA 0,5 ng, 2X Master Mix (One PCR) av 100 mM Tris-HCl (pH 9,1), 0,1 % TritonTMX-100, 200 mM dNTP, 1,5 mM MgCl2, 0,005 U Taq DNA-polymeras och 0,2 µM framåtriktad och bakåtriktad primer med volymavstämning med nukleasfritt vatten. PCR-termocyklern (Thermo Scientific Hybaid Px2) programmerades enligt följande: (1) inledande denaturering i 2 minuter vid 94 ºC i 1 cykel, (2) denaturering vid 94 ºC i 45 s, glödgning vid 54 ºC i 45 s och förlängning vid 72 ºC i 1 minut i 32 cykler, (3) slutlig förlängning vid 72 ºC i 7 minuter. Proverna förvarades vid 4 ºC fram till vidare analys. PCR-produkterna av 16S rRNA (~ 1 500 bp) upptäcktes på 0,8 % agarosgel, renades med hjälp av PCR-produktreningskit (Vivantis, Malaysia), skickades till First Base Co. Ltd. (Malaysia) för DNA-sekvensering. 16S rRNA-gensekvenserna jämfördes sedan med andra sekvenser som fanns tillgängliga i GenBank med hjälp av BLASTN-programmet (Basic Local Alignment Search Tools)10 . Det fylogenetiska trädet konstruerades med hjälp av Muscle-metoden för sekvensanpassning och maximum likelihood-metoden med hjälp av MEGA X med 1 000 replikat av bootstrap-värden11. Alla 16S rRNA-delsekvenser av våra amylasproducerande isolat deponerades i NCBI-databasen.
Bestämning av amylasenzymaktivitet
Metoden följde Bhaskara et al. (2011)12. De tre bakterier som hade de högsta Halo: Koloniförhållandena valdes ut för bestämning av amylasenzymaktivitet. Varje isolat subkulturerades i stärkelsevätskebuljong (g/L): 10,0 löslig stärkelse, 10,0 pepton, 20,0 jästextrakt, 0,05 KH2PO4, 0,015 MnCl2.4H2O, 0,25 MgSO4.7H2O, 0,05 CaCl2.2H2O, 0,01 FeSO4.7H2O och inkuberades vid 37 ºC, 150 rpm i 1, 2 och 3 dagar. Den klara supernatanten (rå extracellulärt amylasenzym) erhölls efter centrifugering vid 10 000 g i 15 minuter vid 4 ºC. Råextraktet koncentrerades med hjälp av MWCO 10 kDa ultracentrifugalproteinkoncentrator (Vivaspin, Sartorius, UK).
DNS-metoden13 användes för att bestämma amylasaktiviteten hos varje bakterieisolat vid varje enzyminduktionsdag. En mL råenzym blandades med 1 mL 1 % stärkelselösning i 1 mL 0,05 M natriumfosfatbuffert pH 7,0. Proverna inkuberades vid 37 °C i 30 minuter. Efter inkubationen tillsattes 0,5 mL DNS-lösning till varje prov för att stoppa reaktionen och därefter kokades vid 100 °C i 5 minuter i vattenbad. Lösningens färgintensitet observerades genom att mäta den optiska densiteten (OD) med en spektrofotometer vid 575 nm. Avläsningen jämfördes med en förberedd blank lösning (utan råenzym). OD-värdena för proverna vid T30min subtraherades från proverna vid T0min eftersom glukos fortfarande fanns kvar i proverna vid T0min efter enzyminduktionsprocessen under 1, 2 och 3 dagar. Processen utfördes i tre exemplar. Koncentrationen av glukos som producerades för varje lösning erhölls från glukosstandardkurvan. Amylasets aktivitet beräknades. En enhet amylasaktivitet definieras som den mängd amylas som krävs för att katalysera bildandet av reducerande socker som motsvarar 1 µmol glukos per minut under analysförhållanden14.
Råenzym från det bakterieisolat som hade den högsta aktiviteten valdes ut för fortsatt arbete.
För att bestämma den specifika enzymaktiviteten hos de utvalda isolaten användes Folin-Lowry-metoden för estimering av totalt protein15. Den specifika enzymaktiviteten mättes med hjälp av följande formel:
Specifik aktivitet (U/mg) = Enzymaktivitet (U/ml)/Extracellulär proteinkoncentration (mg/ml)
Optimalt pH och temperatur för amylasenzymaktivitet
Lösliga stärkelselösningar (1 %) i olika pH från 3 till 10 testades. En mL av olika substratlösningar med 1 % tillsattes tillsammans med 1 mL av respektive buffertar; 0,05 M citratbuffert (pH 3 till 5), 0,05 M natriumfosfatbuffert (pH 6 och 7), 0,05 M Tris-HCl (pH 8 och 9) och 0,05 M glycin NaOH (pH 10). En ml råenzym tillsattes också till dessa buffertar. Proverna inkuberades vid 37 °C i 30 minuter. Den specifika amylasaktiviteten beräknades. Det pH-värde vid vilket den högsta aktiviteten observerades noterades. På samma sätt framställdes olika substratlösningar genom att lösa 1 % löslig stärkelse i pH 7,0-lösningar. 1 mL 1 % löslig stärkelse tillsattes tillsammans med 1 mL 0,05 M natriumfosfatbuffert (pH 7). En mL råenzym tillsattes också till buffertarna. Proverna inkuberades vid 20, 30, 40, 50, 60, 70 och 80 °C i 30 minuter. Den specifika amylasaktiviteten beräknades. Den temperatur vid vilken den högsta aktiviteten observerades noterades. Både optimalt pH och optimal temperatur användes för att bestämma den slutliga specifika amylasenzymaktiviteten.
Statistisk analys
Envägsanalys av varians (One-way ANOVA) användes med Duncan Multiple Range’s Test på SPSS Statistics Ver. 17.0. Resultaten uttrycktes som medelvärde ± SD med statistisk skillnad när p < 0,05.
Resultat
Isolering av amylasproducerande bakterier
I den här studien uppvisade 13 amylaspositiva isolat tydliga zoner på 1 % stärkelseagar med olika Halo : Koloniförhållande. De kolonier som visade tydliga zoner i jodlösning togs som positiva stärkelsenedbrytande bakteriekolonier. Alla bakterieisolat uppvisade liknande kolonimorfologier och visade sig vara grampositiva och stavformade bakterier (tabell 1). Resultatet visade att förhållandet mellan Halo och kolonier varierade mellan 1,18 och 1,71 (tabell 1). Bakterieisolaten användes sedan för att bestämma den optimala induktionstiden för amylasenzym och endast de 3 isolaten med högst specifika aktiviteter visades i 3.4.
Stammeidentifiering av amylasproducerande bakterier
Alla 13 amylaspositiva bakteriestammar genomgick 16S rRNA-gensekvensering för stamidentifiering. BLAST-resultaten visade att alla amylaspositiva isolat tillhör släktet Bacillus, utom ett isolat som tillhör Enterobacter (tabell 2). Deras närmaste släktingar hittades i Indien, Kina, Korea, Indonesien, Argentina, Italien, Israel, USA, Argentina och Sydafrika med ett intervall på 96-100 % identitet.
Fylogenetisk analys
Det fylogenetiska trädet av 13 amylaspositiva bakteriestammar och 2 referensstammar med förmodade amylasenzymer med hjälp av MEGA 7.0 visade att E. cloacae 2.1AL2 skilde sig evolutionärt från de andra Bacillusstammarna (fig. 1). Referensstammen B. thuringiensis serovar konkukian (NCBI accession no. AB617494.1) utvecklades annorlunda än våra Bacillus-bakterier. En annan referensstam B. cereus ATCC 14579 (NCBI accession no. MG708176.1) isolerad från safflorblad, Eqypt, uppvisade dock en liknande utveckling som våra bakterier (fig. 1).
 |
Tabell 1: Egenskaper och Halo : Koloniförhållanden för 13 amylasproducerande bakteriestammar
Klicka här för att visa tabellen |
Optimal amylasenzyminduktionsduration
Det visades att den inkubationsduration som inducerade amylasproduktion med den högsta specifika aktiviteten var 2-3 dagar bland alla 3 isolaten utan statistiska skillnader. Bacillus sp. 3.5AL2 verkade producera den högsta specifika aktiviteten på 0,91 ± 0,12 U/mg vid dag 2 vid 37 °C (tabell 3). Således användes denna stam och detta tillstånd för att inducera amylasproduktion för vidare analys.
Optimalt pH och temperatur för amylasenzymaktivitet
Bacillus sp. 3.5AL2 uppvisade den högsta specifika aktiviteten vid pH 7,0 när 37°C var fixerat och vid 60°C när pH 7,0 var fixerat (fig. 2). Således användes båda de optimala förhållandena för att bestämma specifik amylasaktivitet och 1,97 ± 0,41 U/mg (tabell 4) erhölls.
Tabell 2: Tretton amylaspositiva bakteriestammar som identifierats genom 16S rRNA-analys
| Isolat | Accession
nr. a |
Närmaste släktingb | Accession nr.c | % Identityd | Origine |
| 1.1AL1 | MK578206 | Bacillus pseudomycoides FJAT-hcl-17 | KY653098.1 | 99% | Host Paris polyphylla var. chinensis, Kina |
| 1.1AL2 | MK578207 | Bacillus sp. 188Cu-As | KM349197.1 | 99% | Biofilmer, Argentina |
| 1.2AL3 | MK578208 | Bacillus anthracis SAK4 | MG706137.1 | 100% | Jorden, Republiken Korea |
| 1.4AL1 | MK578209 | Bacillus sp. CNJ732 PL04 | DQ448749.1 | 99% | Marinsediment, USA |
| 1.4AL3 | MK578210 | Bacillus cereus B19 | MK229038.1 | 99% | Vetekorn, Isarael |
| 2.1AL2 | MK578211 | Enterobacter cloacae HNXY160623 | KX431213.1 | 98% | Fågelembryo, Kina |
| 2.3AL1 | MK578212 | Bacillus thuringiensis BD17-E12 | HF584771.1 | 99% | Grapevine root system, Italy |
| 2.3AL8 | MK578213 | Bacillus cereus DFT-1 | KY750685.1 | 98% | Sjövatten från industriområde, Indonesien |
| 2.3AL9 | MK578214 | Bacillus sp. PTP1 | KY910137.1 | 96% | Papaya mealybug tarm, Indien |
| 2.4AL2 | MK578215 | Bacillus pseudomycoides 74 | MH910178.1 | 99% | Indien |
| 3.2AL1 | MK578216 | Bacillus thuringiensis
IARI-IIWP-38 |
KF054891.1 | 99% | Vete rhizospere, Indien |
| 3.4AL1 | MK578217 | Bacillus cereus F4a | MK088302.1 | 99% | Tea rhizosfärjord, Indien |
| 3.5AL2 | MK578218 | Bacillus cereus SP1-AB4 | MH013307.1 | 99% | Marinsvamp, Sydafrika |
aGenBank accession no. av våra stammar deponerade på NCBI:s webbplats (http://www.ncbi.nlm.nih.gov/pubmed)
bNärmaste art med högsta % identitet och högsta Max-poäng vid BLAST-sökning
cGenBank-anslutningsnummer för närmast besläktade stammar på NCBI:s webbplats
dBaserat på resultaten av BLAST-sökningen, identitetsgrad (%) för stammarna jämfört med de
näraste släktingarna.
eBaserat på BLAST-sökresultaten, de närmaste släktingarnas ursprung.
 |
Figur 1: Fylogenetiskt träd av 13 amylaspositiva bakteriestammar och 2 Bacillus-referensstammar
Klicka här för att se bild |
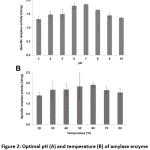 |
Figur 2: Optimalt pH (A) och temperatur (B) för amylasenzymaktivitet från Bacillus sp. 3.5AL2
Klicka här för att visa figur |
Tabell 3: Optimal tid för induktion av amylasenzym
|
Isolat |
Specifikt amylas. aktivitet
(U/mg protein) |
||
| 24 h | 48 h | 72 h | |
| Bacillus sp. 1.1AL2 | 0,21 ± 0,04Ab | 0,40 ± 0,04Ca | 0,41 ± 0,04Ba |
| B. cereus 2.3AL8 | 0,45 ± 0,06Ab | 0,62 ± 0,04Ba | 0,65 ± 0,06Aa |
| Bacillus sp. 3.5AL2 | 0,32 ± 0,11Ab | 0,91 ± 0,12Aa | 0,86 ± 0,11Aa |
Värdena är medelvärden ± SD i tre exemplar.
Kapitala och små bokstäver anger statistiska skillnader (p < 0,05) i kolumnen respektive raden.
Tabell 4: Specifik amylasenzymaktivitet vid optimalt pH och optimal temperatur
| Stam | Aktivitet (U/mg) | Optimal Temp. (°C) | Optimalt pH |
| Bacillus sp. 3.5AL2 | 1,97 ± 0,41 | 60 | 7.0 |
Diskussion
Den thailändska tullens (2017) uppgifter visade att Thailand har importerat industriella enzymer från andra länder till ett värde av 2 625 miljoner Bahts under 2016 och importnivån ökade med 9,40 % per år. Ett av de mest använda enzymerna i Thailand, amylas, har använts inom kläd-, textil- och livsmedelsindustrin. Det saknas dock lokal produktion av kommersiellt amylas och därför syftade detta arbete till att isolera jordbakterier från lokal skog med kapacitet att producera amylas för industriell användning.
Stärkelserika restprodukter kan vara en bättre potentiell källa där amylaspositiva bakterier kan isoleras16. Dessutom kan dessa isoleras från platser som kassavaodlingar, jord och bearbetningsmjölsfabriker17. I detta arbete isolerades 13 amylasproducerande bakterier som identifierades som Bacillus spp. och Enterobacter sp. vilket liknar tidigare rapporter18, 19, 20 där Bacillus spp. oftast påträffades som amylasproducenter.
Våra resultat visade att Bacillus sp. 3.5AL2, isoleringen med den högsta specifika aktiviteten bland de 13 isolaten, producerade den högsta specifika aktiviteten på 0,91 ± 0,12 U/mg vid 2-dagars inkubation vid 37°C. Det optimala pH-värdet och den optimala temperaturen för amylasaktivitet var 7,0 respektive 60 °C. Båda de optimala förhållandena gav en specifik amylasaktivitet från Bacillus sp. 3.5AL2 på 1,97 ± 0,41 U/mg. Det är känt att enzymer som kan arbeta vid hög temperatur anses ha en fördel i industriella processer, t.ex. för att göra stärkelse flytande21. Därför kan amylas från Bacillus sp. 3.5AL2 användas i sådana industrier.
Samma sak gäller för Bacillus sp. GM890, där den optimala temperaturen för amylas var 60 °C20, Bacillus licheniformis AI20, som visade den högsta aktiviteten mellan 60 och 80 °C22 och Bacillus sp. WA21 hade en lägre optimal temperatur på 55 °C för amylas19.
De flesta av de stärkelsenedbrytande bakteriestammarna uppvisade ett pH-värde mellan 6,0 och 7,0 för normal tillväxt och enzymaktivitet4. I detta arbete hade Bacillus sp. 3.5AL2 ett optimalt pH på 7,0, vilket inte kräver någon tillsats av syra eller alkali. Vårt resultat stämmer överens med Bacillus sp. isolerad från dhalindustriavfall som uppvisar en optimal temperatur på 60 °C23 och B. amyloliquefaciens med ett optimalt pH på 7,024. Bacillus sp. WA21 uppvisade dock ett optimalt pH för amylaset på 6,0, vilket är lägre än vad som konstaterades i denna studie19.
Den här typen av studie är mycket viktig för startpunkten för kommersiell amylasproduktion i Thailand. Ytterligare forskningsutredningar är dock nödvändiga för att göra amylasproduktionen kostnadseffektiv. Det finns ett behov av grundlig enzymkarakterisering, ytterligare bestämning av termostabilitet, pH-stabilitet, påverkan av olika metalljoner och olika substrat, särskilt jordbruksavfall i Thailand, och deras koncentrationer.
Efter att ha erhållit alla data som behövs enligt ovan, tror man att produktionen av kommersiellt amylas i Thailands enzymindustrier kommer att öka och spridas i varje region och i sin tur kommer att gynna landets ekonomi på grund av att man är självförsörjande på sina egna resurser för att producera amylasenzym.
Slutsats
Detta är den första rapporten om identifiering av 13 amylasproducerande bakterieisolat från jord i Nasinuan Community Forest, Maha Sarakham. Alla bakterier identifierades som Bacillus spp. utom en från Enterobacter-släktet. Dessa bakterier kan användas för produktion av amylas och kan i framtiden användas lokalt och nationellt inom jordbruket, livsmedelsindustrin och textilindustrin. Detta kommer således att minska kostnaderna för import av industriella enzymer från andra länder, ge en hållbar lokal enzymproduktion och förbättra landets ekonomi.
Acknowledgments
Författarna vill tacka Mahasarakham University (Grant Year 2018) för finansiellt stöd som tilldelats SD, och Faculty of Technology, Mahasarakham University (Grant Year 2017) som tilldelats VL.
Interessentkonflikter
Författarna förklarar att de inte har några intressekonflikter.
- Pandey A. Glucoamylase research: En översikt. 47:439-445 (1995).
- Burhan A.I., Nisa U., Gokhan C., Omer C., Ashabil A. och Osman G. Enzymatiska egenskaper hos ett nytt termostabilt, termofilt, alkaliskt och chelatorresistent amylas från ett alkalifilt Bacillus-isolat ANT-6. Process Biochem. 38: 1397-1403 (2003).
- Gurung N., Ray S., Bose S., and Rai, V.A. Broader view: Mikrobiella enzymer och deras betydelse för industrier, medicin och andra områden. Biomed Res Internat. 1-18. doi:10.1155/2013/ 329121 (2013).
- Gupta R., Gigras P., Mohapatra H., Goswami V.K. och Chauhan B. Microbial α-amylases: a biotechnological perspective. Process Biochem. 38:1599-1616 (2003).
- Takasaki Y. Ett amylas som producerar maltotetros och från maltopentos från circulans. Agric Biol Chem. 47:2193-2199 (1983).
- Fogarty W.M., and Kelly C.T. Amylase, amyloglucosidase and related glucanases. Rose A.H. Economic Microbiology, Microbial Enzymes and Bioconversion, New York Academic Press Inc, vol. 5. Pp. 115-170 (1980).
- Wind R.D., Buitelaar R.M.G., Huizing H.J., and Dijkhuizen L. Characterization of a new Bacillus stearothermophilus isolate: a highly thermostable α-amylase producing strain. Appl Microbiol Biotechnol. 41:155-162 (1994).
- Brumm P.J., Hebeda R.E. och Teague W.M. Purification & characterization of commercialized, cloned megaterium α-amylase. Del I: rening & hydrolytiska egenskaper. Stärkelse. 43:319-323 (1991).
- Takasaki Y. Ett amylas som producerar maltotrios från subtilis. Agric Bio. Chem. 49:1091-1097 (1985).
- Lowry O.H., Rosebrough N.J., and Randall R.J. Protein measurement with the folin phenol reagents. J Biol Chem. 193: 265-275 (1951).
- Altschul S.F., Gish W., Miller W., Myers E.W. och Lipman D.J. Basic local alignment search tool. J Mol 215:403-410 (1990).
- Bhaskara R.K.V., Ashwini K., Gaurav K. och Karthik L. Optimering, produktion och partiell rening av extracellulärt α-amylas från Bacillus marini. Arc Appl Sci Res. 3:33-42 (2011).
- GhoseK. Mätning av cellulaseaktiviteter. Pure Appl Chem. 59: 257-268 (1987).
- Haq U.I., Ashraf H., Iqbal J. och Qadeer M.A. Produktion av α-amylas av Bacillus licheniformis med hjälp av ett ekonomiskt medium. Biores Technol. 87:57-61 (2003).
- Mishra S. och Behera N. Amylasaktivitet hos stärkelsenedbrytande bakterier isolerade från jord som tar emot köksavfall. Afr J Biotechnol. 7: 3326-3331 (2008).
- Fossi B.T., Taveaand F., and Ndjonenkeu T. Production and Partial Characterization of a Themostable amylase from Ascomycetes yeast strain isolated from starchy soils. Afr J Biotechnol. 4:14-18 (2005).
- Cordeiro C.A.M. Production and propertiesof α-amylase from thermophilic Bacillus sp. Brazil J Microbiol. 33:57-61 (2002).
- Asad W. Extracellular enzyme production by indigenous thermophilic bacteria: Partiell rening och karakterisering av α-amylas från Bacillus sp. WA21. Pakistan J Botany. 43:1045-1052 (2011).
- Kim T.U. Purification and characterization of a maltotetraose-forming alkaline α-amylase from an alkalophilic Bacillus strain, GM8901. Appl Envin Microbiol. 61: 83105-3112 (1995).
- Sivaramakrishnan S., Gangadharan D., Madhavan K.N. och Pandey A. Fast odling av Bacillus amyloliquefaciens för produktion av alfa-amylas. Food Technol 44:269-274 (2006).
- Abdel-Fattah Y.R., Soliman N.A., El-Toukhy N.M., El-Gendi H., and Ahmed R.S. Purification, and characterization of thermostable α-amylase produced by Bacillus licheniformis isolate AI20. J Chem. 2013:1-11 (2013).
- Thippeswamy S., Girigowda K., and Mulimani V.H. Isolation and identification of α-amylase producing Bacillus from Dhal
industry waste. Indian J Biochem Biophysics. 43: 295-298 (2006). - Abd-ElhalemT., El-Sawy M., Rawia F.G. och Khadiga A.A.T. Produktion av amylas från Bacillus amyloliquefaciens under submergerad jäsning med hjälp av vissa agroindustriella biprodukter. Ann Agri Sci. 60:193-202 (2015).
- Tamura K., and Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol Biol Evol. 10:512-526 (1993).

(Besökt 12 737 gånger, 21 besök idag).