Manuscript accepted on :29 Aug 19
Published online on: 18-09-2019 Plagiarism Check: Yes
Reviewed by: Farooq Sher
Second Review by: Grigorios Kyriakopoulos
Final Approval by: Ayush Dogra
Jak Citeclose | Publication Historyclose
Vijitra Luang-In1* , Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 i Kedsukon Maneewan1
, Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 i Kedsukon Maneewan1
1Natural Antioxidant Innovation Research Unit, Department of Biotechnology, Faculty of Technology, Mahasarakham University, Khamriang, Kantarawichai, Maha Sarakham, 44150, Thailand.
2International College of Digital Innovation, Chiang Mai University, Chiang Mai 50200, Thailand.
Corresponding Author E-mail: [email protected]
DOI : https://dx.doi.org/10.13005/bpj/1735
Abstract
This study aimed to isolate and identify bacteria that can produce amylase enzyme from the unexplored Nasinuan Forest, Kantarawichai District, Mahasarakham Province, Thailand. Trzynaście izolatów bakterii o zdolności do produkcji amylazy na 1% agarze skrobiowym zostało zidentyfikowanych przy użyciu sekwencjonowania 16S rRNA. Dwanaście bakterii było gram-dodatnich, o kształcie pręcika i zidentyfikowano je jako Bacillus spp., a jedna bakteria o charakterze gram-ujemnym i kształcie pręcika to Enterobacter cloacae. Ich najbliżsi krewni występowali w Indiach, Chinach, Korei, Indonezji, Argentynie, Włoszech, Izraelu, USA, Argentynie i RPA. Bakterie te badano pod kątem specyficznej aktywności amylazy po 1-3 dniowej indukcji enzymatycznej 1% skrobią w temp. 37°C. Wyniki wykazały najwyższą aktywność specyficzną w 2. dniu inkubacji w kolejności: Bacillus cereus 3.5AL2 > 3.4AL1 > 1.4AL3 i dlatego do dalszych analiz wybrano 2-dniową indukcję enzymatyczną. Bacillus sp. 3.5AL2 wykazał najwyższą specyficzną aktywność enzymatyczną amylazy 1.97 ± 0.41 U/mg białka w optymalnych warunkach 60°C i pH 7.0 po 30 min inkubacji z 1% skrobią w 0.05 M buforze PBS. Ten szczep bakterii produkujący amylazę oferuje duży potencjał do zastosowań w przemyśle spożywczym i rolniczym w Tajlandii.
Słowa kluczowe
Amylaza; Bakteria; las Nasinuan; gleba
Pobierz ten artykuł jako:![]()
Copy the following to cite this article:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3).
Copy the following to cite this URL:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3). Available from: http://biomedpharmajournal.org/?p=28497
Introduction
Amylazy hydrolityczne skrobi (α-amylaza, β-amylaza i glukoamylaza) są jednymi z najszerzej stosowanych enzymów w dzisiejszej biotechnologii. Glukoamylazy (glukanohydrolaza 1,4-α-D-glukanu; EC 3.2.1.3) lub amyloglukozydazy, to amylazy działające egzoaktywnie, które wytwarzają glukozę z nieredukującego końca skrobi i odpowiednich oligosacharydów. Glukoamylazy są stosowane komercyjnie do przekształcania malto-oligosacharydów w glukozę1. Chociaż amylazy są produkowane z różnych źródeł (mikroorganizmów, roślin i zwierząt), amylazy pochodzące z mikrobów są najbardziej odpowiednie do produkcji przemysłowej ze względu na krótki okres wzrostu mikroorganizmów, niskie koszty produkcji, przyjazne dla środowiska zachowanie, mniej problemów z obsługą przez pracowników, wydajność2 i łatwość manipulacji genami bakterii3. Bakterie i grzyby mają tendencję do wydzielania amylaz na zewnątrz komórek w celu przeprowadzenia pozakomórkowego trawienia skrobi na cukry. Rosnące zapotrzebowanie przemysłu na amylazy mikrobiologiczne obserwuje się ze względu na ich specyficzność reakcji, łagodne warunki niezbędne do przeprowadzenia reakcji oraz mniejsze zużycie energii niż w przypadku konwencjonalnych nieenzymatycznych metod chemicznych.
Extensive application of amylase in food, starch liquefaction, saccharification, brewing, detergent, paper, textile and distilling industries, has brought about a greater attention for the increase in the indigenous production of α-amylase4. Bacillus jest powszechnym źródłem bakterii do przemysłowej produkcji amylazy. Jednakże, różne szczepy mają różne optymalne warunki wzrostu i profil produkcji enzymatycznej. Według doniesień, szczepy Bacillus były szeroko wykorzystywane przemysłowo do produkcji α-amylazy, w tym B. amyloliquefaciens, B. subtilis5, B. licheniformis6, B. stearothermophilus7, B. megaterium8 i B. circulans9.
Do tej pory udokumentowano szereg bakterii wytwarzających amylazę, jednak nie przeprowadzono badań nad bakteriami wytwarzającymi amylazę z gleby w Nasinuan Community Forest, Kantharawichai District, Mahasarakham Province, Thailand. Las ten wydaje się być bogaty w różnorodność biologiczną mikroorganizmów, które mogą być przydatne w produkcji enzymów przemysłowych, w tym amylazy. Jest to pierwszy raport identyfikujący bakterie produkujące amylazę wyizolowane z lasu Nasinuan. Te bakteryjne amylazy mogą być lokalnie stosowane w różnych gałęziach przemysłu, w tym w przemyśle celulozowo-papierniczym, tekstylnym, bioetanolu, browarnictwie, przetwórstwie żywności, paszach dla zwierząt i rolnictwie w Tajlandii w przyszłości.
Materiały i metody
Próbki gleby
Próbki gleby zostały losowo zebrane poniżej powierzchni gleby 15 cm i przechowywane w torbach styropianowych z Nasinuan Community Forest, Kantarawichai District, Mahasarakham Province, Thailand (powierzchnia 9.6 ha; współrzędne 16.340941, 103.210799).
Isolacja bakterii wytwarzających amylazę
Próbkę gleby (10 g) zawieszono w 90 mL sterylnego 0,85% roztworu NaCl. Zawiesiny (100 µL) w seryjnych rozcieńczeniach rozprowadzono na agarze skrobiowym (g/L); 3,0 ekstrakt wołowy, 10,0 skrobia rozpuszczalna, 15,0 agar pH 7,0 i inkubowano w temperaturze 37 ºC przez 7 dni. Następnie płytki zalewano jodyną Grama. Wszystkie kolonie z wytworzeniem wyraźnej strefy wokół kolonii posiewano do płynnego bulionu i co najmniej pięciokrotnie wykonywano posiewy w celu uzyskania czystych izolatów, co potwierdzano barwieniem metodą Grama i 1000-krotną obserwacją mikroskopową. Czyste izolaty inokulowano punktowo na agar skrobiowy i inkubowano w temperaturze 37°C przez 7 dni. Średnice wyraźnych stref nad średnicami kolonii mierzono przy użyciu linijki jako stosunek Halo : Kolonia.
Sekwencjonowanie genu 16S rRNA i analiza filogenetyczna
Do identyfikacji czystych izolatów bakteryjnych wykorzystano genomowe DNA uzyskane powyższą metodą oraz uniwersalne startery: starter forward 27F 5′-GAGAGTTGATYCTGCTCAG-3′ i starter reverse 1492R 5’AAGGAGGTGATCCARCCGCA -3′. W 25 µL mieszaniny PCR znajdowało się genomowe DNA 0,5 ng, 2X Master Mix (One PCR) o składzie 100 mM Tris-HCl (pH 9,1), 0,1% TritonTMX-100, 200 mM dNTP, 1,5 mM MgCl2, 0,005 U Taq DNA Polymerase oraz 0,2 µM primera forward i reverse z uzupełnieniem objętości wodą wolną od nukleaz. Termocykler PCR (Thermo Scientific Hybaid Px2) zaprogramowano w następujący sposób: (1) wstępna denaturacja przez 2 min w temp. 94 ºC przez 1 cykl; (2) denaturacja w temp. 94 ºC przez 45 s; annealing w temp. 54 ºC przez 45 s i przedłużenie w temp. 72 ºC przez 1 min przez 32 cykle; (3) końcowe przedłużenie w temp. 72 ºC przez 7 min. Próbki przechowywano w temperaturze 4 ºC do czasu dalszej analizy. Produkty PCR 16S rRNA (~ 1500 bp) wykrywano na 0,8% żelu agarozowym, oczyszczano przy użyciu zestawu do oczyszczania produktów PCR (Vivantis, Malezja), przesyłano do First Base Co. Ltd. (Malezja) do sekwencjonowania DNA. Następnie sekwencje genu 16S rRNA porównano z innymi dostępnymi w GenBank przy użyciu programu BLASTN (Basic Local Alignment Search Tools)10. Drzewo filogenetyczne zostało skonstruowane przy użyciu metody Muscle’a dla wyrównania sekwencji oraz metody największego prawdopodobieństwa przy użyciu programu MEGA X z 1000 replikami wartości bootstrapowych11. Wszystkie sekwencje częściowe 16S rRNA naszych izolatów wytwarzających amylazę zostały zdeponowane w bazie danych NCBI.
Określanie aktywności enzymatycznej amylazy
Metoda była zgodna z Bhaskara et al. (2011)12. Trzy bakterie o najwyższym stosunku Halo: Kolonia zostały wybrane do oznaczenia aktywności enzymu amylazy. Każdy izolat był subkulturowany w płynnym bulionie skrobiowym (g/L): 10.0 skrobia rozpuszczalna, 10.0 pepton, 20.0 ekstrakt drożdżowy, 0.05 KH2PO4, 0.015 MnCl2.4H2O, 0.25 MgSO4.7H2O, 0.05 CaCl2.2H2O, 0.01 FeSO4.7H2O i inkubowany w 37 ºC, 150 rpm przez 1, 2 i 3 dni. Klarowny supernatant (surowy enzym amylazy zewnątrzkomórkowej) uzyskano po odwirowaniu przy 10 000g przez 15 min w temp. 4ºC. Surowy ekstrakt zatężano przy użyciu ultrakoncentratora białkowego MWCO 10 kDa (Vivaspin, Sartorius, UK).
Do oznaczenia aktywności amylazy każdego izolatu bakteryjnego w poszczególnych dniach indukcji enzymatycznej zastosowano metodę DNS13. Jeden mL surowego enzymu mieszano z 1 mL 1% roztworu skrobi w 1 mL 0,05 M buforu fosforanu sodu o pH 7,0. Próbki inkubowano w temperaturze 37°C przez 30 min. Po inkubacji do każdej próbki dodawano 0,5 mL roztworu DNS w celu zatrzymania reakcji, a następnie gotowano w łaźni wodnej w temperaturze 100°C przez 5 min. Intensywność barwy roztworu obserwowano mierząc gęstość optyczną (OD) za pomocą spektrofotometru przy długości fali 575 nm. Odczyt porównywano z przygotowanym roztworem ślepym (bez surowego enzymu). Wartości OD próbek w czasie T30min odejmowano od wartości próbek w czasie T0min, ponieważ glukoza nadal pozostawała w próbkach w czasie T0min po procesie indukcji enzymatycznej w czasie 1, 2, 3 dni. Proces ten przeprowadzono w trzech powtórzeniach. Stężenie glukozy wytworzonej dla każdego roztworu uzyskano z krzywej wzorcowej glukozy. Obliczono aktywność amylazy. Jedną jednostkę aktywności amylazy definiuje się jako ilość amylazy potrzebną do katalizowania powstawania cukru redukującego, która jest równa 1 µmol glukozy na min w warunkach oznaczania14.
Do dalszych prac wybrano surowy enzym izolatu bakteryjnego o najwyższej aktywności.
W celu określenia specyficznej aktywności enzymatycznej wybranych izolatów zastosowano metodę Folina-Lowry’ego do szacowania białka całkowitego15. Aktywność specyficzną enzymu mierzono według następującego wzoru.
Aktywność specyficzna (U/mg) = Aktywność enzymu (U/ml)/Stężenie białka zewnątrzkomórkowego (mg/ml)
Optymalne pH i temperatura aktywności enzymatycznej amylazy
Przebadano roztwory skrobi rozpuszczalnej (1%) o różnym pH od 3 do 10. Jeden mL różnych 1% roztworów substratów dodano wraz z 1 mL odpowiednich buforów: 0,05 M buforu cytrynianowego (pH 3 do 5), 0,05 M buforu fosforanu sodu (pH 6 i 7), 0,05 M Tris-HCl (pH 8 i 9) i 0,05 M glicyny NaOH (pH 10). Do tych buforów dodawano również 1 ml surowego enzymu. Próbki inkubowano w temperaturze 37°C przez 30 min. Obliczono aktywność specyficzną amylazy. Odnotowano pH, przy którym obserwowano najwyższą aktywność. Podobnie wykonano różne roztwory substratów poprzez rozpuszczenie 1% rozpuszczalnej skrobi w roztworach o pH 7,0. Jeden mL 1% rozpuszczalnej skrobi dodano wraz z 1 mL 0,05 M buforu fosforanu sodu (pH 7). Do buforów dodano również 1 mL surowego enzymu. Próbki inkubowano w temperaturze 20, 30, 40, 50, 60, 70 i 80°C przez 30 min. Obliczono specyficzną aktywność amylazy. Odnotowano temperaturę, w której zaobserwowano najwyższą aktywność. Zarówno optymalne pH, jak i temperatura zostały użyte do określenia ostatecznej specyficznej aktywności enzymu amylazy.
Analiza statystyczna
Jednoczynnikowa analiza wariancji (One-way ANOVA) została użyta z testem Duncana dla wielu zakresów w SPSS Statistics Ver. 17.0. Wyniki wyrażono jako średnie ± SD z różnicą statystyczną gdy p < 0,05.
Wyniki
Isolacja bakterii wytwarzających amylazę
W tym badaniu, 13 izolatów amylazododatnich wykazało wyraźne strefy na 1% agarze skrobiowym z różnymi stosunkami Halo : Kolonie. Kolonie wykazujące wyraźne strefy w roztworze jodyny zostały uznane za pozytywne kolonie bakterii rozkładających skrobię. Wszystkie izolaty bakteryjne wykazywały podobną morfologię kolonii i występowały jako bakterie Gram-dodatnie o kształcie pręcików (Tabela 1). Stosunek Halo : Kolonia wynosił od 1,18 do 1,71 (Tabela 1). Izolaty bakteryjne zostały następnie użyte do określenia optymalnego czasu indukcji enzymu amylazy i tylko 3 izolaty o najwyższej specyficznej aktywności zostały przedstawione w 3.4.
Identyfikacja szczepów bakterii wytwarzających amylazę
Wszystkie 13 amylazododatnich szczepów bakteryjnych zostało poddanych sekwencjonowaniu genu 16S rRNA w celu identyfikacji szczepu. Wyniki badania metodą BLAST wykazały, że wszystkie izolaty amylazododatnie należą do rodzaju Bacillus, z wyjątkiem jednego izolatu należącego do Enterobacter (Tabela 2). Ich najbliższych krewnych znaleziono w Indiach, Chinach, Korei, Indonezji, Argentynie, Włoszech, Izraelu, USA, Argentynie i Południowej Afryce z zakresem 96-100% identyczności.
Analiza filogenetyczna
Drzewo filogenetyczne 13 amylazododatnich szczepów bakteryjnych i 2 szczepów referencyjnych z putatywnymi enzymami amylazowymi przy użyciu programu MEGA 7.0 wykazało, że E. cloacae 2.1AL2 ewolucyjnie różnił się od pozostałych szczepów Bacillus (Rys. 1). Referencyjny szczep B. thuringiensis serovar konkukian (NCBI accession no. AB617494.1) ewoluował inaczej niż nasze bakterie Bacillus. Natomiast inny szczep referencyjny B. cereus ATCC 14579 (NCBI accession no. MG708176.1) wyizolowany z liści krokosza barwierskiego, Eqypt wykazywał podobną ewolucję do naszych bakterii (Rys. 1).
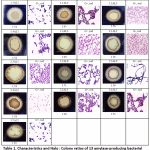 |
Table 1: Characteristics and Halo : Colony ratios of 13 amylase-producing bacterial strains
Click here to View table |
Optimal amylase enzyme induction duration
Wykazano, że czas trwania inkubacji, który indukował produkcję amylazy o najwyższej specyficznej aktywności wynosił 2-3 dni wśród wszystkich 3 izolatów bez różnic statystycznych. Bacillus sp. 3.5AL2 wydawał się wytwarzać najwyższą aktywność specyficzną na poziomie 0,91 ± 0,12 U/mg w 2. dniu inkubacji w temperaturze 37°C (Tabela 3). Tak więc, ten szczep i te warunki zostały wykorzystane do indukowania produkcji amylazy do dalszych analiz.
Optymalne pH i temperatura aktywności enzymu amylazy
Bacillus sp. 3.5AL2 wykazał najwyższą specyficzną aktywność w pH 7.0, gdy 37°C było ustalone i w 60°C, gdy pH 7.0 było ustalone (Rys. 2). Tak więc, oba optymalne warunki zostały użyte do określenia specyficznej aktywności amylazy i uzyskano 1,97 ± 0,41 U/mg (Tabela 4).
Tabela 2: Trzynaście amylazododatnich szczepów bakteryjnych zidentyfikowanych przez analizę 16S rRNA
| Izolat | Accession
no. a |
Closest relativeb | Accession no.c | % Identityd | Origine |
| 1.1AL1 | MK578206 | Bacillus pseudomycoides FJAT-hcl-17 | KY653098.1 | 99% | Host Paris polyphylla var. chinensis, China |
| 1.1AL2 | MK578207 | Bacillus sp. 188Cu-As | KM349197.1 | 99% | Biofilmy, Argentyna |
| 1.2AL3 | MK578208 | Bacillus anthracis SAK4 | MG706137.1 | 100% | Soil, Republic of Korea |
| 1.4AL1 | MK578209 | Bacillus sp. CNJ732 PL04 | DQ448749.1 | 99% | Marine sediment, USA |
| 1.4AL3 | MK578210 | Bacillus cereus B19 | MK229038.1 | 99% | Ziarno pszenicy, Isarael |
| 2.1AL2 | MK578211 | Enterobacter cloacae HNXY160623 | KX431213.1 | 98% | Avian embryo, China |
| 2.3AL1 | MK578212 | Bacillus thuringiensis BD17-E12 | HF584771.1 | 99% | Grapevine root system, Italy |
| 2.3AL8 | MK578213 | Bacillus cereus DFT-1 | KY750685.1 | 98% | Seawater of industrial area, Indonesia |
| 2.3AL9 | MK578214 | Bacillus sp. PTP1 | KY910137.1 | 96% | Papaya mealybug gut, India |
| 2.4AL2 | MK578215 | Bacillus pseudomycoides 74 | MH910178.1 | 99% | Indie |
| 3.2AL1 | MK578216 | Bacillus thuringiensis
IARI-IIWP-38 |
KF054891.1 | 99% | Ryzosfera pszenicy, Indie |
| 3.4AL1 | MK578217 | Bacillus cereus F4a | MK088302.1 | 99% | Tea rhizosphere soil, India |
| 3.5AL2 | MK578218 | Bacillus cereus SP1-AB4 | MH013307.1 | 99% | Gąbka morska, RPA |
aGenBank accession no. of our strains deposited on NCBI website (http://www.ncbi.nlm.nih.gov/pubmed)
bClosest species with highest % identity and highest Max score on BLAST search
cGenBank accession no. of closest relative strains on NCBI website
dBased on BLAST search results, identity (%) of strains compared to the
closest relatives.
eBased on BLAST search results, origin of the closest relatives.
 |
Figure 1: Phylogenetic tree of 13 amylase-positive bacterial strains and 2 Bacillus reference strains
Click here to View figure |
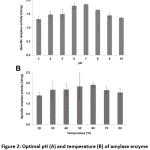 |
Figure 2: Optimal pH (A) and temperature (B) of amylase enzyme activity from Bacillus sp. 3.5AL2
Click here to View figure |
Tabela 3: Optymalny czas indukcji enzymu amylazy
|
Izolat |
Specyficzna aktywność amylazy aktywność
(U/mg białka) |
||
| 24 h | 48 h | 72 h | |
| Bacillus sp. 1.1AL2 | 0,21 ± 0,04Ab | 0,40 ± 0,04Ca | 0,41 ± 0,04Ba |
| B. cereus 2.3AL8 | 0,45 ± 0,06Ab | 0,62 ± 0,04Ba | 0,65 ± 0,06Aa |
| Bacillus sp. 3.5AL2 | 0,32 ± 0,11Ab | 0,91 ± 0,12Aa | 0,86 ± 0,11Aa |
Values are means ± SD in triplicate.
Duże i małe litery oznaczają różnice statystyczne (p < 0,05) odpowiednio w kolumnie i wierszu.
Tabela 4: Aktywność specyficzna enzymu amylazy przy optymalnym pH i temperaturze
| Szczep | Aktywność (U/mg) | Optymalna Temp. (°C) | Optimal pH |
| Bacillus sp. 3.5AL2 | 1.97 ± 0.41 | 60 | 7.0 |
Dyskusja
Dane tajskiego urzędu celnego (2017) wykazały, że Tajlandia importowała enzymy przemysłowe z innych krajów o wartości 2,625 mln bahtów w 2016 r., a wskaźnik importu wzrósł o 9,40% rocznie. Jeden z najszerzej stosowanych enzymów w Tajlandii, amylaza, został zastosowany w przemyśle odzieżowym, tekstylnym i spożywczym. Brakuje jednak lokalnej produkcji komercyjnej amylazy i dlatego niniejsza praca miała na celu wyizolowanie bakterii glebowych z lokalnego lasu ze zdolnością do produkcji amylazy do zastosowań przemysłowych.
Resztki bogate w skrobię mogą być lepszym potencjalnym źródłem, w którym można wyizolować bakterie amylazododatnie16. Ponadto, mogą one być izolowane z miejsc takich jak farmy manioku, gleba i fabryki mąki przetwórczej17. W tej pracy wyizolowano 13 bakterii wytwarzających amylazę i zidentyfikowano je jako Bacillus spp. i Enterobacter sp., co jest podobne do wcześniejszych doniesień18, 19, 20, gdzie Bacillus spp. były najczęściej znajdowane jako producenci amylazy.
Nasze wyniki wykazały, że Bacillus sp. 3.5AL2, izolat o najwyższej specyficznej aktywności spośród 13 izolatów, wytwarzał najwyższą specyficzną aktywność na poziomie 0,91 ± 0,12 U/mg w 2-dniowej inkubacji w 37°C. Optymalne pH i temperatura dla aktywności amylazy wynosiły odpowiednio 7,0 i 60°C. W obu optymalnych warunkach uzyskano specyficzną aktywność amylazy z Bacillus sp. 3.5AL2 na poziomie 1,97 ± 0,41 U/mg. Wiadomo, że enzymy, które mogą pracować w wysokiej temperaturze są uważane za mające przewagę w procesach przemysłowych, takich jak upłynnianie skrobi21. Tak więc amylaza z Bacillus sp. 3.5AL2 ma potencjalne zastosowanie w takich gałęziach przemysłu.
Podobnie, Bacillus sp. GM890 wykazał optymalną temperaturę dla amylazy w 60°C20, Bacillus licheniformis AI20 wykazał najwyższą aktywność w zakresie 60-80°C22, a Bacillus sp. WA21 miał niższą optymalną temperaturę 55°C dla amylazy19.
Większość szczepów bakterii degradujących skrobię wykazywała zakres pH pomiędzy 6,0 a 7,0 dla normalnego wzrostu i aktywności enzymatycznej4. Podobnie, w tej pracy, Bacillus sp. 3.5AL2 miał optymalne pH 7.0, które nie wymaga żadnego dodatku kwasu lub zasady. Nasz wynik był zgodny z wyizolowanym z odpadów przemysłu dhal Bacillus sp. wykazującym optymalną temperaturę 60°C23 oraz B. amyloliquefaciens o optymalnym pH 7,024. Niemniej jednak Bacillus sp. WA21 wykazał optymalne pH amylazy na poziomie 6,0, które jest niższe niż to stwierdzone w tym badaniu19.
Tego rodzaju badanie jest bardzo ważne dla punktu wyjścia komercyjnej produkcji amylazy w Tajlandii. Jednak dodatkowe badania są niezbędne, aby produkcja amylazy stała się opłacalna. Istnieje potrzeba dokładnej charakterystyki enzymu, dalszego określenia termostabilności, stabilności pH, wpływu różnych jonów metali i różnych substratów, zwłaszcza odpadów rolniczych w Tajlandii, oraz ich stężeń.
Po uzyskaniu wszystkich potrzebnych danych, jak wspomniano powyżej, uważa się, że produkcja komercyjnej amylazy w przemyśle enzymatycznym Tajlandii będzie wzrastać i rozpowszechniać się w każdym regionie, a z kolei będzie przynosić korzyści gospodarce kraju ze względu na samowystarczalność w zakresie własnych zasobów do produkcji enzymu amylazy.
Wniosek
Jest to pierwsze sprawozdanie z identyfikacji 13 izolatów bakterii wytwarzających amylazę z gleby w Nasinuan Community Forest, Maha Sarakham. Wszystkie bakterie zostały zidentyfikowane jako Bacillus spp. z wyjątkiem jednej z rodzaju Enterobacter. Bakterie te mogą być wykorzystane do produkcji amylazy i zastosowane w przyszłości na poziomie lokalnym i krajowym w rolnictwie, przetwórstwie żywności i przemyśle tekstylnym. Zmniejszy to koszty importu enzymów przemysłowych z innych krajów, zapewni trwałość lokalnej produkcji enzymów i wzmocni gospodarkę kraju.
Podziękowania
Autorzy chcieliby podziękować Mahasarakham University (Grant Year 2018) za wsparcie finansowe przyznane SD, oraz Faculty of Technology, Mahasarakham University (Grant Year 2017) przyznany VL.
Conflict of Interest
Autorzy deklarują brak konfliktu interesów.
- Pandey A. Glucoamylase research: An overview. 47:439-445 (1995).
- Burhan A.I., Nisa U., Gokhan C., Omer C., Ashabil A., and Osman G. Enzymatic properties of a novel thermostable, thermophilic, alkaline and chelator resistant amylase from an alkaliphilic Bacillus isolate ANT-6. Process Biochem. 38: 1397-1403 (2003).
- Gurung N., Ray S., Bose S., and Rai, V.A. Broader view: Enzymy mikrobiologiczne i ich znaczenie w przemyśle, medycynie i poza nią. Biomed Res Internat. 1-18. doi:10.1155/2013/ 329121 (2013).
- Gupta R., Gigras P., Mohapatra H., Goswami V.K., and Chauhan B. Microbial α-amylases: a biotechnological perspective. Process Biochem. 38:1599-1616 (2003).
- Takasaki Y. An amylase producing maltotetrose and from maltopentose from circulans. Agric Biol Chem. 47:2193-2199 (1983).
- Fogarty W.M., and Kelly C.T. Amylase, amyloglucosidase and related glucanases. Rose A.H. Economic Microbiology, Microbial Enzymes and Bioconversion, New York Academic Press Inc, vol. 5. Pp. 115-170 (1980).
- Wind R.D., Buitelaar R.M.G., Huizing H.J., and Dijkhuizen L. Characterization of a new Bacillus stearothermophilus isolate: a highly thermostable α-amylase producing strain. Appl Microbiol Biotechnol. 41:155-162 (1994).
- Brumm P.J., Hebeda R.E., and Teague W.M. Purification & characterization of commercialized, cloned megaterium α-amylase. Część I: oczyszczanie & właściwości hydrolityczne. Starch. 43:319-323 (1991).
- Takasaki Y. Amylaza produkująca maltotriozę z subtilis. Agric Bio. Chem. 49:1091-1097 (1985).
- Lowry O.H., Rosebrough N.J., and Randall R.J. Protein measurement with the folin phenol reagents. J Biol Chem. 193: 265-275 (1951).
- Altschul S.F., Gish W., Miller W., Myers E.W., and Lipman D.J. Basic local alignment search tool. J Mol 215:403-410 (1990).
- Bhaskara R.K.V., Ashwini K., Gaurav K., and Karthik L. Optimization, production and partial purification of extracellular α-amylase from Bacillus marini. Arc Appl Sci Res. 3:33-42 (2011).
- GhoseK. Measurement of cellulase activities. Pure Appl Chem. 59: 257-268 (1987).
- Haq U.I., Ashraf H., Iqbal J., and Qadeer M.A. Production of α-amylase by Bacillus licheniformis using an economical medium. Biores Technol. 87:57-61 (2003).
- Mishra S., and Behera N. Amylase activity of starch degrading bacteria is isolated from soil receiving kitchen wastes. Afr J Biotechnol. 7: 3326-3331 (2008).
- Fossi B.T., Taveaand F., and Ndjonenkeu T. Production and Partial Characterization of a Themostable amylase from Ascomycetes yeast strain isolated from starchy soils. Afr J Biotechnol. 4:14-18 (2005).
- Cordeiro C.A.M. Production and propertiesof α-amylase from thermophilic Bacillus sp. Brazil J Microbiol. 33:57-61 (2002).
- Asad W. Extracellular enzyme production by indigenous thermophilic bacteria: Partial purification and characterization of α-amylase by Bacillus sp. WA21. Pakistan J Botany. 43:1045-1052 (2011).
- Kim T.U. Purification and characterization of a maltotetraose-forming alkaline α-amylase from an alkalophilic Bacillus strain, GM8901. Appl Envin Microbiol. 61: 83105-3112 (1995).
- Sivaramakrishnan S., Gangadharan D., Madhavan K.N., and Pandey A. Solid culturing of Bacillus amyloliquefaciens for alpha amylase production. Food Technol 44:269-274 (2006).
- Abdel-Fattah Y.R., Soliman N.A., El-Toukhy N.M., El-Gendi H., and Ahmed R.S. Purification, and characterization of thermostable α-amylase produced by Bacillus licheniformis isolate AI20. J Chem. 2013:1-11 (2013).
- Thippeswamy S., Girigowda K., and Mulimani V.H. Isolation and identification of α-amylase producing Bacillus from Dhal
industry waste. Indian J Biochem Biophysics. 43: 295-298 (2006). - Abd-ElhalemT., El-Sawy M., Rawia F.G., and Khadiga A.A.T. Production of amylase from Bacillus amyloliquefaciens under submerged fermentation using some agro-industrialby-products. Ann Agri Sci. 60:193-202 (2015).
- Tamura K., and Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol Biol Evol. 10:512-526 (1993).

(Visited 12,737 times, 21 visits today)