Manuscript geaccepteerd op :29 aug 19
Publicished online on: 18-09-2019 Plagiaatcontrole: Ja
Reviewed by: Farooq Sher
Tweede Review door: Grigorios Kyriakopoulos
Final Approval by: Ayush Dogra
Hoe te Citeclose | Publication Historyclose
Vijitra Luang-In1* , Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 en Kedsukon Maneewan1
, Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 en Kedsukon Maneewan1
1Natural Antioxidant Innovation Research Unit, Department of Biotechnology, Faculty of Technology, Mahasarakham University, Khamriang, Kantarawichai, Maha Sarakham, 44150, Thailand.
2International College of Digital Innovation, Chiang Mai University, Chiang Mai 50200, Thailand.
Corresponderende auteur E-mail: [email protected]
DOI : https://dx.doi.org/10.13005/bpj/1735
Abstract
Deze studie had tot doel bacteriën te isoleren en te identificeren die amylase-enzym kunnen produceren uit het onontgonnen Nasinuan Forest, Kantarawichai District, Mahasarakham Province, Thailand. Dertien bacteriële isolaten met een amylase-producerende capaciteit op 1% zetmeelagar werden geïdentificeerd met behulp van 16S rRNA-sequencing. Twaalf bacteriën waren gram-positief, staafvormig en geïdentificeerd als Bacillus spp. en één bacterie met gram-negatief en staafvormig karakter was Enterobacter cloacae. Hun naaste verwanten werden gevonden in India, China, Korea, Indonesië, Argentinië, Italië, Israël, de VS en Zuid-Afrika. Deze bacteriën werden getest op specifieke amylase-activiteit na 1-3 dagen enzyminductie met 1% zetmeel bij 37°C. De resultaten toonden de hoogste specifieke activiteit op dag 2 incubatie in de volgorde: Bacillus cereus 3.5AL2 > 3.4AL1 > 1.4AL3 en dus werd 2-daagse enzyminductie gekozen voor verdere analyse. Bacillus sp. 3.5AL2 bleek de hoogste specifieke amylase enzymactiviteit te vertonen van 1.97 ± 0.41 U/mg eiwit bij de optimale condities van 60°C en pH 7.0 na 30 min incubatie met 1% zetmeel in 0.05 M PBS buffer. Deze amylase-producerende bacteriestam biedt een groot potentieel voor toepassingen in de voedsel- en landbouwindustrie in Thailand.
Keywords
Amylase; Bacteriën; Nasinuan bos; bodem
Download dit artikel als:![]()
Copy the following to cite this article:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3).
Copy the following to cite this URL:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3). Available from: http://biomedpharmajournal.org/?p=28497
Introductie
De zetmeelhydrolytische amylasen (α-amylase, β-amylase en glucoamylase) zijn een van de meest gebruikte enzymen in de hedendaagse biotechnologie. Glucoamylasen (1,4-α-D-glucanglucanohydrolase; EC 3.2.1.3) of amyloglucosidasen, zijn exo-actieven amylasen die glucose produceren uit het niet-reducerende uiteinde van zetmeel en de overeenkomstige oligosacchariden. Glucoamylasen worden commercieel toegepast voor de omzetting van malto-oligosacchariden in glucose1. Hoewel amylasen uit verschillende bronnen (micro-organismen, planten en dieren) worden geproduceerd, zijn amylasen uit microben het meest geschikt voor industriële productie vanwege de korte groeiperiode van micro-organismen, de lage productiekosten, het milieuvriendelijke gedrag, de geringe hanteerbaarheid voor werknemers, de productiviteit2 en de gemakkelijke manipulatie van bacteriële genen3. Bacteriën en schimmels hebben de neiging amylasen buiten de cellen af te scheiden om zetmeel extra cellulair in suikers te verteren. Er is een toenemende industriële vraag naar microbiële amylasen vanwege hun specifieke reactievermogen, de milde omstandigheden die nodig zijn voor de reactie en het lagere energieverbruik dan bij de conventionele niet-enzymatische chemische methoden.
De uitgebreide toepassing van amylase in de voedingsmiddelen-, zetmeelcondensatie-, versuikering-, brouwerij-, detergenten-, papier-, textiel- en distillatie-industrie heeft geleid tot een grotere aandacht voor de toename van de inheemse productie van α-amylase4. Bacillus is een veelgebruikte bacteriële bron voor de industriële productie van amylase. Verschillende stammen hebben echter verschillende optimale groeiomstandigheden en enzymatisch productieprofiel. Naar verluidt zijn Bacillus-stammen op grote schaal industrieel gebruikt om α-amylase te produceren, waaronder B. amyloliquefaciens, B. subtilis5, B. licheniformis6, B. stearothermophilus7, B. megaterium8 en B. circulans9.
Tot op heden is een aantal amylase-producerende bacteriën gedocumenteerd, maar er is geen studie gerapporteerd over amylase-producerende bacteriën uit de bodem in het Nasinuan Community Forest, Kantharawichai District, Mahasarakham Province, Thailand. Dit bos lijkt rijk te zijn aan microbiële biodiversiteit die nuttig kan zijn voor de productie van industriële enzymen, waaronder amylase. Dit is het eerste verslag over de identificatie van amylase-producerende bacteriën die uit Nasinuan Forest zijn geïsoleerd. Deze bacteriële amylases kunnen in de toekomst lokaal worden toegepast in verschillende industrieën, waaronder pulp en papier, textiel, bio-ethanol, brouwerij, voedselverwerking, diervoeder en landbouw in Thailand.
Materialen en Methoden
Bodemmonsters
Bodemmonsters werden willekeurig verzameld onder het bodemoppervlak 15 cm en bewaard in polystyreen zakken uit Nasinuan Community Forest, Kantarawichai District, Mahasarakham Provincie, Thailand (oppervlakte van 9.6 hectare; coördinaat van 16.340941, 103.210799).
Ontdekking van amylase-producerende bacteriën
Bodemmonster (10 g) werd gesuspendeerd in 90 mL steriele 0,85% NaCl-oplossing. De suspensies (100 µL) van seriële verdunningen werden uitgespreid op zetmeelagar (g/L); 3.0 runderextract, 10.0 oplosbaar zetmeel, 15.0 agar pH 7.0 en geïncubeerd bij 37 ºC gedurende 7 dagen. Vervolgens werden de platen overspoeld met Gram-jodium. Kolonies met een duidelijke zone rond de kolonies werden ondergekweekt in vloeibare bouillon en ten minste vijf keer gestreept om zuivere isolaten te verkrijgen, bevestigd door Gramkleuring en microscopische observatie met 1000X-licht. De zuivere isolaten werden geënt op zetmeelagar en gedurende 7 dagen geïncubeerd bij 37°C. De diameters van de heldere zones over de diameters van de kolonies werden gemeten met een liniaal als de Halo : Kolonie verhouding.
16S rRNA-gensequencing en fylogenetische analyse
Pure bacteriële isolaten werden geïdentificeerd met behulp van genomisch DNA verkregen met de bovenstaande methode en universele primers: forward primer 27F 5′-GAGAGTTGATYCTGGCTCAG-3′ en reverse primer 1492R 5’AAGGAGGTGATCCARCCGCA -3′. In 25 µL PCR-mengsel was het samengesteld uit genomisch DNA 0,5 ng, 2X Master Mix (One PCR) van 100 mM Tris-HCl (pH 9,1), 0,1% TritonTMX-100, 200 mM dNTP, 1,5 mM MgCl2, 0,005 U Taq DNA Polymerase en 0,2 µM voorwaartse en achterwaartse primer met volumeaanpassing met nucleasevrij water. De PCR-thermocycler (Thermo Scientific Hybaid Px2) werd als volgt geprogrammeerd: (1) initiële denaturatie gedurende 2 min bij 94 ºC gedurende 1 cyclus; (2) denaturatie bij 94 ºC gedurende 45 s; annealing bij 54 ºC gedurende 45 s, en extensie bij 72 ºC gedurende 1 min gedurende 32 cycli; (3) laatste extensie bij 72 ºC gedurende 7 min. De monsters werden tot de verdere analyse bij 4 ºC bewaard. De PCR-producten van 16S rRNAs (~ 1.500 bp) werden gedetecteerd op 0,8% agarose gel, gezuiverd met de PCR-productzuiveringskit (Vivantis, Maleisië), verzonden naar First Base Co. Ltd. (Maleisië) voor DNA-sequentiebepaling. De 16S rRNA-gensequenties werden vervolgens vergeleken met andere in GenBank beschikbare sequenties met behulp van het BLASTN-programma (Basic Local Alignment Search Tools)10. De fylogenetische boom werd geconstrueerd met behulp van de Muscle-methode voor sequentie-uitlijning en de maximum likelihood-methode met behulp van MEGA X met 1.000 replicaten van bootstrap-waarden11. Alle 16S rRNA partiële sequenties van onze amylase-producerende isolaten werden gedeponeerd op NCBI-databank.
Bepaling van de amylase-enzymactiviteit
De methode volgde Bhaskara et al. (2011)12. De drie bacteriën met de hoogste Halo: Kolonie-verhoudingen werden gekozen voor de bepaling van de amylase-enzymactiviteit. Elk isolaat werd ondergekweekt in een vloeibare zetmeelbouillon (g/L): 10,0 oplosbaar zetmeel, 10,0 pepton, 20,0 gistextract, 0,05 KH2PO4, 0,015 MnCl2.4H2O, 0,25 MgSO4.7H2O, 0,05 CaCl2.2H2O, 0,01 FeSO4.7H2O en geïncubeerd bij 37 ºC, 150 rpm gedurende 1, 2 en 3 dagen. Het heldere supernatant (ruw extracellulair amylase-enzym) werd verkregen na centrifugatie bij 10.000g gedurende 15 min bij 4 ºC. Het ruwe extract werd geconcentreerd met behulp van een MWCO 10 kDa ultracentrifuge eiwitconcentrator (Vivaspin, Sartorius, UK).
De DNS-methode13 werd gebruikt om de amylaseactiviteit van elk bacterie-isolaat te bepalen op elke enzym-inductiedag. Eén ml ruw enzym werd gemengd met 1 ml zetmeeloplossing van 1% in 1 ml 0,05 M natriumfosfaatbuffer pH 7,0. De monsters werden gedurende 30 minuten bij 37°C geïncubeerd. Na incubatie werd 0,5 mL DNS-oplossing toegevoegd aan elk monster om de reactie te stoppen en vervolgens gedurende 5 minuten bij 100°C gekookt in het waterbad. De kleurintensiteit van de oplossing werd waargenomen door met een spectrofotometer de optische dichtheid (OD) bij 575 nm te meten. De afgelezen waarde werd vergeleken met een bereide blanco-oplossing (zonder ruw enzym). De OD-waarden van monsters op T30min werden afgetrokken van die van monsters op T0min, aangezien in de monsters op T0min na de enzyminductie op 1, 2 en 3 dagen nog glucose aanwezig was. Het proces werd in drievoud uitgevoerd. De glucoseconcentratie die voor elke oplossing werd geproduceerd, werd verkregen uit de glucose-standaardcurve. De activiteit van amylase werd berekend. Een eenheid amylaseactiviteit wordt gedefinieerd als de hoeveelheid amylase die nodig is om de vorming van reducerende suiker te katalyseren, wat gelijk is aan 1 µmol glucose per minuut onder testomstandigheden14.
Het ruwe enzym van het bacterie-isolaat met de hoogste activiteit werd gekozen voor verder werk.
Om de specifieke enzymactiviteit van de geselecteerde isolaten te bepalen, werd de Folin-Lowry-methode voor de schatting van het totale eiwit gebruikt15. De specifieke enzymactiviteit werd gemeten met de volgende formule.
Specifieke activiteit (U/mg) = Enzymactiviteit (U/ml)/Extracellulaire eiwitconcentratie (mg/ml)
Optimale pH en temperatuur van amylase-enzymactiviteit
Oplosbare zetmeeloplossingen (1%) met verschillende pH van 3 tot 10 werden getest. Een mL van verschillende 1% substraatoplossing werd toegevoegd samen met 1 mL van de respectieve buffers; 0,05 M citraatbuffer (pH 3 tot 5), 0,05 M natriumfosfaatbuffer (pH 6 en 7), 0,05 M Tris-HCl (pH 8 en 9) en 0,05 M glycine NaOH (pH 10). Aan deze buffers werd eveneens 1 ml ruw enzym toegevoegd. De monsters werden gedurende 30 minuten bij 37°C geïncubeerd. De specifieke amylaseactiviteit werd berekend. De pH waarbij de hoogste activiteit werd waargenomen, werd genoteerd. Evenzo werden verschillende substraatoplossingen gemaakt door 1% oplosbaar zetmeel op te lossen in pH 7,0-oplossingen Eén mL oplosbaar zetmeel van 1% werd toegevoegd samen met 1 mL 0,05 M natriumfosfaatbuffer (pH 7). Aan de buffers werd ook 1 ml ruw enzym toegevoegd. De monsters werden gedurende 30 minuten geïncubeerd bij 20, 30, 40, 50, 60, 70 en 80°C. De specifieke amylaseactiviteit werd berekend. De temperatuur waarbij de hoogste activiteit werd waargenomen, werd genoteerd. Zowel optimale pH als temperatuur werden gebruikt om de uiteindelijke specifieke amylase enzymactiviteit te bepalen.
Statistische analyse
One-Way Analysis Of Variance (One-way ANOVA) werd gebruikt met Duncan Multiple Range’s Test op SPSS Statistics Ver. 17.0. Resultaten werden uitgedrukt als gemiddelden ± SD met statistisch verschil wanneer p < 0.05.
Resultaten
Isolatie van amylase-producerende bacteriën
In deze studie vertoonden 13 amylase-positieve isolaten duidelijke zones op 1% zetmeel agar met verschillende Halo : Kolonie verhoudingen. De kolonies die duidelijke zones van jodiumoplossing vertoonden, werden genomen als positieve zetmeelafbrekende bacteriële kolonies. Alle bacteriële isolaten vertoonden een soortgelijke koloniemorfologie en bleken Gram-positieve en staafvormige bacteriën te zijn (tabel 1). Het resultaat toonde Halo : Kolonie verhoudingen variërend van 1,18 tot 1,71 (tabel 1). Bacteriële isolaten werden vervolgens gebruikt om de optimale duur van amylase-enzyminductie te bepalen en alleen de 3 isolaten met de hoogste specifieke activiteiten werden getoond in 3.4.
Stamidentificatie van amylase-producerende bacteriën
Alle 13 amylase-positieve bacteriestammen werden onderworpen aan 16S rRNA-gensequencing voor stamidentificatie. De BLAST resultaten toonden aan dat alle amylase-positieve isolaten behoren tot het genus Bacillus, behalve één isolaat dat behoort tot Enterobacter (Tabel 2). Hun naaste verwanten werden gevonden in India, China, Korea, Indonesië, Argentinië, Italië, Israël, VS en Zuid-Afrika met een range van 96-100% identiteit.
Phylogenetische analyse
De fylogenetische boom van 13 amylase-positieve bacteriestammen en 2 referentiestammen met putatieve amylase enzymen met behulp van MEGA 7.0 toonde aan dat E. cloacae 2.1AL2 evolutionair verschillend was van de andere Bacillus stammen (Fig. 1). De B. thuringiensis serovar konkukian (NCBI accession no. AB617494.1) referentiestam evolueerde anders dan onze Bacillus bacteriën. Een andere referentiestam B. cereus ATCC 14579 (NCBI-toetredingsnr. MG708176.1), geïsoleerd uit saffloerblad, Eqypt, vertoonde echter een soortgelijke evolutie als onze bacteriën (Fig. 1).
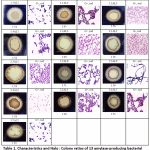 |
Tabel 1: Kenmerken en Halo : Colony Ratios of 13 amylase-producing bacterial strains
Klik hier om de tabel te bekijken |
Optimale duur voor de inductie van amylase-enzymen
Er werd aangetoond dat de incubatieduur die amylaseproductie induceerde met de hoogste specifieke activiteit 2-3 dagen was bij alle 3 isolaten zonder statistische verschillen. Bacillus sp. 3.5AL2 leek de hoogste specifieke activiteit te produceren met 0,91 ± 0,12 U/mg op dag 2 bij 37°C (Tabel 3). Daarom werd deze stam en deze conditie gebruikt voor het induceren van amylaseproductie voor verdere analyse.
Optimale pH en temperatuur van amylase-enzymactiviteit
Bacillus sp. 3.5AL2 vertoonde de hoogste specifieke activiteit bij pH 7,0 wanneer 37°C werd vastgesteld en bij 60°C wanneer pH 7,0 werd vastgesteld (Fig. 2). Aldus werden beide optimale omstandigheden gebruikt om de specifieke amylaseactiviteit te bepalen en werd 1,97 ± 0,41 U/mg (tabel 4) verkregen.
Tabel 2: Dertien amylase-positieve bacteriestammen geïdentificeerd aan de hand van 16S rRNA-analyse
| Isolaat | Accessie
nr. a |
Nauwst verwanteb | Accessienr.c | % Identityd | Origine |
| 1.1AL1 | MK578206 | Bacillus pseudomycoides FJAT-hcl-17 | KY653098.1 | 99% | Host Paris polyphylla var. chinensis, China |
| 1.1AL2 | MK578207 | Bacillus sp. 188Cu-As | KM349197.1 | 99% | Biofilms, Argentinië |
| 1.2AL3 | MK578208 | Bacillus anthracis SAK4 | MG706137.1 | 100% | Bodem, Republiek Korea |
| 1.4AL1 | MK578209 | Bacillus sp. CNJ732 PL04 | DQ448749.1 | 99% | Marien sediment, USA |
| 1.4AL3 | MK578210 | Bacillus cereus B19 | MK229038.1 | 99% | Wit graan, Isarael |
| 2.1AL2 | MK578211 | Enterobacter cloacae HNXY160623 | KX431213.1 | 98% | Avian embryo, China |
| 2.3AL1 | MK578212 | Bacillus thuringiensis BD17-E12 | HF584771.1 | 99% | Grapevine wortelstelsel, Italië |
| 2.3AL8 | MK578213 | Bacillus cereus DFT-1 | KY750685.1 | 98% | Zeewater van industriegebied, Indonesië |
| 2.3AL9 | MK578214 | Bacillus sp. PTP1 | KY910137.1 | 96% | Papaya wolluisdarm, India |
| 2.4AL2 | MK578215 | Bacillus pseudomycoides 74 | MH910178.1 | 99% | India |
| 3.2AL1 | MK578216 | Bacillus thuringiensis
IARI-IIWP-38 |
KF054891.1 | 99% | Tarwegries, India |
| 3.4AL1 | MK578217 | Bacillus cereus F4a | MK088302.1 | 99% | Tea rhizosphere soil, India |
| 3.5AL2 | MK578218 | Bacillus cereus SP1-AB4 | MH013307.1 | 99% | Marine sponge, South Africa |
aGenBank accession no. van onze stammen gedeponeerd op de NCBI-website (http://www.ncbi.nlm.nih.gov/pubmed)
bNabijste soort met hoogste %-identiteit en hoogste Max-score bij BLAST-zoekopdracht
GenBank-toetredingsnr. van nauwst verwante stammen op NCBI-website
dBaseerd op BLAST-zoekresultaten, identiteit (%) van stammen vergeleken met de
nabijste verwanten.
eOp basis van BLAST-zoekresultaten, herkomst van de meest verwante stammen.
| Figuur 1: Fylogenetische boom van 13 amylase-positieve bacteriestammen en 2 Bacillus-referentiestammen | Figuur 1. Fylogenetische boom van 13 amylase-positieve bacteriestammen en 2 Bacillus-referentiestammen |
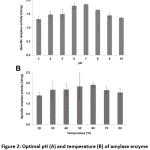 |
Figuur 2: Optimale pH (A) en temperatuur (B) van de amylase-enzymactiviteit van Bacillus sp. 3.5AL2
Klik hier om figuur te bekijken |
Tabel 3: Optimale duur van de inductie van het amylase-enzym
|
Isolaat |
Specifieke amylase activiteit
(U/mg eiwit) |
||
| 24 h | 48 h | 72 h | |
| Bacillus sp. 1.1AL2 | 0.21 ± 0.04Ab | 0.40 ± 0.04Ca | 0.41 ± 0.04Ba |
| B. cereus 2.3AL8 | 0.45 ± 0.06Ab | 0.62 ± 0.04Ba | 0.65 ± 0.06Aa |
| Bacillus sp. 3.5AL2 | 0,32 ± 0,11Ab | 0,91 ± 0,12Aa | 0,86 ± 0,11Aa |
Waarden zijn gemiddelden ± SD in drievoud.
Hoofdletters en kleine letters wijzen op statistische verschillen (p < 0,05) in de kolom en de rij, respectievelijk.
Tabel 4: Specifieke amylase-enzymactiviteit bij optimale pH en temperatuur
| Stam | Activiteit (U/mg) | Optimale Temp. (°C) | Optimale pH |
| Bacillus sp. 3.5AL2 | 1.97 ± 0.41 | 60 | 7.0 |
Discussie
De gegevens van de Thaise douane (2017) toonden aan dat Thailand in 2016 industriële enzymen uit andere landen heeft ingevoerd met een waarde van 2.625 miljoen Bahts en dat het invoertempo met 9,40% per jaar is gestegen. Een van de meest gebruikte enzymen in Thailand, amylase, is toegepast in de kleding-, textiel- en voedingsmiddelenindustrie. Er is echter een gebrek aan lokale productie van commercieel amylase en daarom was dit werk gericht op het isoleren van bodembacteriën uit het lokale bos met de capaciteit om amylase te produceren voor industrieel gebruik.
Zetmeelrijke residuen kunnen een betere potentiële bron zijn waar amylase-positieve bacteriën kunnen worden geïsoleerd16. Bovendien zouden deze kunnen worden geïsoleerd uit plaatsen zoals cassaveboerderijen, de bodem en verwerkende meelfabrieken17. In dit werk werden 13 amylase-producerende bacteriën geïsoleerd en geïdentificeerd als Bacillus spp. en Enterobacter sp. wat vergelijkbaar is met de eerdere rapporten18, 19, 20 waar Bacillus spp. meestal werden gevonden als amylaseproducenten.
Onze resultaten toonden aan dat Bacillus sp. 3.5AL2, het isolaat met de hoogste specifieke activiteit van de 13 isolaten, de hoogste specifieke activiteit produceerde van 0,91 ± 0,12 U/mg bij 2-daagse incubatie bij 37°C. De optimale pH en temperatuur voor amylaseactiviteit waren respectievelijk 7,0 en 60°C. Beide optimale omstandigheden gaven de specifieke amylaseactiviteit van Bacillus sp. 3.5AL2 van 1,97 ± 0,41 U/mg. Het is bekend dat enzymen die bij hoge temperatuur kunnen werken, worden beschouwd als een voordeel bij industriële processen zoals de vloeibaarmaking van zetmeel21. Amylase van Bacillus sp. 3.5AL2 heeft dus een potentiële toepassing in dergelijke industrieën.
Ook Bacillus sp. GM890 vertoonde de optimale temperatuur voor amylase bij 60°C20, Bacillus licheniformis AI20 vertoonde de hoogste activiteit tussen 60-80°C22 en Bacillus sp. WA21 had een lagere optimale temperatuur van 55°C voor amylase19.
De meeste zetmeel-afbrekende bacteriestammen toonden een pH-bereik tussen 6,0 en 7,0 voor normale groei en enzymactiviteit4. Ook in dit werk had Bacillus sp. 3.5AL2 een optimale pH-waarde van 7,0, waarvoor geen zuur of base hoefde te worden toegevoegd. Onze bevinding was in overeenstemming met Bacillus sp. geïsoleerd uit afval van de dhal-industrie die een optimale temperatuur van 60°C23 vertoonde en B. amyloliquefaciens met een optimale pH van 7,024. Niettemin vertoonde Bacillus sp. WA21 een optimale pH van het amylase van 6,0, wat minder is dan in deze studie gevonden19.
Dit soort studie is zeer belangrijk voor het startpunt van commerciële amylase productie in Thailand. Aanvullend onderzoek is echter essentieel om amylase productie kosteneffectief te maken. Er is behoefte aan een grondige karakterisering van het enzym, verdere bepaling van de thermostabiliteit, pH-stabiliteit, invloed van verschillende metaalionen en verschillende substraten, vooral landbouwafval in Thailand, en hun concentraties.
Na het verkrijgen van alle benodigde gegevens zoals hierboven vermeld, wordt gedacht dat de productie van commercieel amylase in Thailand enzymenindustrieën zal toenemen en wijdverspreid zal zijn in elke regio en op zijn beurt de economie van het land ten goede zal komen als gevolg van zelf-afhankelijkheid van zijn eigen middelen om amylase-enzym te produceren.
Conclusie
Dit is het eerste rapport van de identificatie van 13 amylase-producerende bacteriële isolaten uit de bodem in Nasinuan Community Forest, Maha Sarakham. Alle bacteriën werden geïdentificeerd als Bacillus spp. behalve één van Enterobacter genus. Deze bacteriën kunnen worden gebruikt voor de productie van amylase en in de toekomst lokaal en nationaal worden toegepast in de landbouw, de voedselverwerkende industrie en de textielindustrie. Dit zal dus de kosten van industriële enzymimport uit andere landen verminderen, duurzaamheid van lokale enzymproductie bieden en de economie van de natie verbeteren.
Acknowledgments
De auteurs willen Mahasarakham University (Grant Year 2018) bedanken voor financiële steun toegekend aan SD, en Faculty of Technology, Mahasarakham University (Grant Year 2017) toegekend aan VL.
Conflict of Interest
De auteurs verklaren geen belangenconflict.
- Pandey A. Glucoamylase onderzoek: An overview. 47:439-445 (1995).
- Burhan A.I., Nisa U., Gokhan C., Omer C., Ashabil A., and Osman G. Enzymatic properties of a novel thermostable, thermophilic, alkaline and chelator resistant amylase from an alkaliphilic Bacillus isolate ANT-6. Process Biochem. 38: 1397-1403 (2003).
- Gurung N., Ray S., Bose S., and Rai, V.A. Broader view: Microbial enzymen and their relevance in industries, medicine and beyond. Biomed Res Internat. 1-18. doi:10.1155/2013/ 329121 (2013).
- Gupta R., Gigras P., Mohapatra H., Goswami V.K., and Chauhan B. Microbial α-amylases: a biotechnological perspective. Process Biochem. 38:1599-1616 (2003).
- Takasaki Y. An amylase producing maltotetrose and from maltopentose from circulans. Agric Biol Chem. 47:2193-2199 (1983).
- Fogarty W.M., and Kelly C.T. Amylase, amyloglucosidase and related glucanases. Rose A.H. Economic Microbiology, Microbial Enzymes and Bioconversion, New York Academic Press Inc, vol. 5. Pp. 115-170 (1980).
- Wind R.D., Buitelaar R.M.G., Huizing H.J., and Dijkhuizen L. Characterization of a new Bacillus stearothermophilus isolate: a highly thermostable α-amylase producing strain. Appl Microbiol Biotechnol. 41:155-162 (1994).
- Brumm P.J., Hebeda R.E., and Teague W.M. Purification & characterization of commercialized, cloned megaterium α-amylase. Part I: purification & hydrolytic properties. Zetmeel. 43:319-323 (1991).
- Takasaki Y. An amylase producing maltotriose from subtilis. Agric Bio. Chem. 49:1091-1097 (1985).
- Lowry O.H., Rosebrough N.J., and Randall R.J. Protein measurement with the folin phenol reagents. J Biol Chem. 193: 265-275 (1951).
- Altschul S.F., Gish W., Miller W., Myers E.W., and Lipman D.J. Basic local alignment search tool. J Mol 215:403-410 (1990).
- Bhaskara R.K.V., Ashwini K., Gaurav K., and Karthik L. Optimization, production and partial purification of extracellular α-amylase from Bacillus marini. Arc Appl Sci Res. 3:33-42 (2011).
- GhoseK. Meting van cellulase activiteiten. Pure Appl Chem. 59: 257-268 (1987).
- Haq U.I., Ashraf H., Iqbal J., and Qadeer M.A. Production of α-amylase by Bacillus licheniformis using an economical medium. Biores Technol. 87:57-61 (2003).
- Mishra S., and Behera N. Amylase activity of starch degrading bacteria is isolated from soil receiving kitchen wastes. Afr J Biotechnol. 7: 3326-3331 (2008).
- Fossi B.T., Taveaand F., and Ndjonenkeu T. Production and Partial Characterization of a Themostable amylase from Ascomycetes yeast strain isolated from starchy soils. Afr J Biotechnol. 4:14-18 (2005).
- Cordeiro C.A.M. Production and propertiesof α-amylase from thermophilic Bacillus sp. Brazil J Microbiol. 33:57-61 (2002).
- Asad W. Extracellulaire enzymproductie door inheemse thermofiele bacteriën: Gedeeltelijke zuivering en karakterisering van α-amylase door Bacillus sp. WA21. Pakistan J Botany. 43:1045-1052 (2011).
- Kim T.U. Purification and characterization of a maltotetraose-forming alkaline α-amylase from an alkalophilic Bacillus strain, GM8901. Application Envin Microbiol. 61: 83105-3112 (1995).
- Sivaramakrishnan S., Gangadharan D., Madhavan K.N., and Pandey A. Solid culturing of Bacillus amyloliquefaciens for alpha amylase production. Food Technol 44:269-274 (2006).
- Abdel-Fattah Y.R., Soliman N.A., El-Toukhy N.M., El-Gendi H., and Ahmed R.S. Purification, and characterization of thermostable α-amylase produced by Bacillus licheniformis isolate AI20. J Chem. 2013:1-11 (2013).
- Thippeswamy S., Girigowda K., and Mulimani V.H. Isolation and identification of α-amylase producing Bacillus from Dhal
industry waste. Indian J Biochem Biophysics. 43: 295-298 (2006). - Abd-ElhalemT., El-Sawy M., Rawia F.G., and Khadiga A.A.T. Production of amylase from Bacillus amyloliquefaciens under submerged fermentation using some agro-industrialby-products. Ann Agri Sci. 60:193-202 (2015).
- Tamura K., and Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol Biol Evol. 10:512-526 (1993).

(12.737 keer bekeken, 21 bezoeken vandaag)