Manoscritto accettato il :29 Ago 19
Pubblicato online il: 18-09-2019 Controllo del plagio: Sì
Recensito da: Farooq Sher
Seconda recensione di: Grigorios Kyriakopoulos
Approvazione finale di: Ayush Dogra
Come Citeclose | Publication Historyclose
Vijitra Luang-In1* , Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 e Kedsukon Maneewan1
, Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 e Kedsukon Maneewan1
1Natural Antioxidant Innovation Research Unit, Dipartimento di Biotecnologia, Facoltà di Tecnologia, Mahasarakham University, Khamriang, Kantarawichai, Maha Sarakham, 44150, Thailandia.
2International College of Digital Innovation, Chiang Mai University, Chiang Mai 50200, Thailandia.
Corresponding Author E-mail: [email protected]
DOI : https://dx.doi.org/10.13005/bpj/1735
Abstract
Questo studio ha mirato a isolare e identificare i batteri che possono produrre l’enzima amilasi dalla foresta inesplorata Nasinuan, Kantarawichai District, Mahasarakham Province, Thailand. Tredici isolati batterici con capacità di produrre amilasi su agar di amido all’1% sono stati identificati usando il sequenziamento del 16S rRNA. Dodici batteri erano gram-positivi, a forma di asta e identificati come Bacillus spp. e un batterio con carattere gram-negativo e a forma di asta era Enterobacter cloacae. I loro parenti più prossimi sono stati trovati in India, Cina, Corea, Indonesia, Argentina, Italia, Israele, USA, Argentina e Sud Africa. Questi batteri sono stati testati per l’attività amilasica specifica dopo 1-3 giorni di induzione enzimatica con 1% di amido a 37°C. I risultati hanno mostrato la più alta attività specifica al secondo giorno di incubazione nell’ordine: Bacillus cereus 3.5AL2 > 3.4AL1 > 1.4AL3 e quindi l’induzione enzimatica di 2 giorni è stata scelta per ulteriori analisi. Bacillus sp. 3.5AL2 ha mostrato la più alta attività enzimatica amilasica specifica di 1,97 ± 0,41 U/mg di proteina alle condizioni ottimali di 60°C e pH 7,0 dopo 30 minuti di incubazione con 1% di amido in tampone PBS 0,05 M. Questo ceppo batterico produttore di amilasi offre un grande potenziale per le applicazioni nelle industrie alimentari e agricole in Thailandia.
Keywords
Amilasi; Batteri; foresta Nasinuan; suolo
Scarica questo articolo come:![]()
Copia quanto segue per citare questo articolo:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolamento e identificazione di batteri produttori di amilasi dal suolo in Nasinuan Community Forest, Maha Sarakham, Thailandia. Biomed Pharmacol J 2019;12(3).
Copy the following to cite this URL:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3). Disponibile da: http://biomedpharmajournal.org/?p=28497
Introduzione
Le amilasi idrolitiche dell’amido (α-amilasi, β-amilasi e glucoamilasi) sono uno degli enzimi più utilizzati nella biotecnologia attuale. Le glucoamilasi (1,4-α-D-glucano glucanoidrolasi; EC 3.2.1.3) o amiloglucosidasi, sono amilasi ad azione esterna che producono glucosio dall’estremità non riducente dell’amido e dagli oligosaccaridi corrispondenti. Le glucoamilasi sono implementate commercialmente per la trasformazione di malto-oligosaccaridi in glucosio1. Anche se le amilasi sono prodotte da diverse fonti (microrganismi, piante e animali), le amilasi dai microbi sono più adatti per la produzione industriale a causa del breve periodo di crescita microbica, produzione a basso costo efficace, comportamento ecologico, meno problemi di gestione per i lavoratori, produttività2 e facile manipolazione dei geni batterici3. Batteri e funghi tendono a secernere amilasi al di fuori delle cellule per eseguire la digestione extracellulare di amido in zuccheri. La crescente domanda industriale di amilasi microbiche è stata osservata a causa della loro specificità di reazione, condizioni miti prerequisito per la reazione, e meno consumo di energia rispetto ai metodi chimici non enzimatici convenzionali.
L’applicazione estensiva di amilasi nel cibo, liquefazione dell’amido, saccarificazione, produzione di birra, detergenti, carta, tessile e industrie di distillazione, ha portato a una maggiore attenzione per l’aumento della produzione indigena di α-amilasi4. Bacillus è una fonte batterica comune per la produzione industriale di amilasi. Tuttavia, diversi ceppi hanno diverse condizioni di crescita ottimale e profilo di produzione enzimatica. Secondo quanto riferito, i ceppi di Bacillus sono stati ampiamente utilizzati a livello industriale per produrre α-amilasi tra cui B. amyloliquefaciens, B. subtilis5, B. licheniformis6, B. stearothermophilus7, B. megaterium8 e B. circulans9.
Fino ad oggi, è stato documentato un certo numero di batteri produttori di amilasi, tuttavia, non è stato riportato alcuno studio sui batteri produttori di amilasi dal suolo della foresta comunitaria Nasinuan, distretto Kantharawichai, provincia di Mahasarakham, Thailandia. Questa foresta sembra essere ricca di biodiversità microbica che può essere utile nella produzione di enzimi industriali tra cui l’amilasi. Questo è il primo rapporto per identificare i batteri produttori di amilasi isolati dalla foresta di Nasinuan. Queste amilasi batteriche possono essere applicate localmente a varie industrie tra cui cellulosa e carta, tessile, bioetanolo, birreria, trasformazione alimentare, alimentazione animale e agricoltura in Thailandia in futuro.
Materiali e metodi
Campioni di suolo
I campioni di suolo sono stati raccolti a caso sotto la superficie del suolo di 15 cm e conservati in sacchetti di polistirolo da Nasinuan Community Forest, Kantarawichai District, Mahasarakham Province, Thailandia (area di 9.6 ettari; coordinate di 16.340941, 103.210799).
Isolamento dei batteri produttori di amilasi
Il campione di suolo (10 g) è stato sospeso in 90 mL di soluzione sterile di NaCl 0,85%. Le sospensioni (100 µL) di diluizioni seriali sono state stese su agar amido (g/L); 3.0 estratto di manzo, 10.0 amido solubile, 15.0 agar pH 7.0 e incubate a 37 ºC per 7 giorni. Successivamente, le piastre sono state inondate con iodio di Gram. Tutte le colonie con formazione di una zona chiara intorno alle colonie sono state subcoltivate in brodo liquido e striate almeno cinque volte per ottenere isolati puri come confermato dalla colorazione di Gram e dall’osservazione al microscopio ottico 1000X. Gli isolati puri sono stati inoculati su agar di amido e incubati a 37°C per 7 giorni. I diametri delle zone chiare rispetto ai diametri delle colonie sono stati misurati usando un righello come rapporto Halo : Colonia.
Il sequenziamento del gene 16S rRNA e l’analisi filogenetica
Gli isolati batterici puri sono stati identificati utilizzando i DNA genomici ottenuti con il metodo sopra descritto e i primer universali: primer forward 27F 5′-GAGAGTTTGATYCTGGCTCAG-3′ e primer reverse 1492R 5’AAGGAGGTGATCCARCCGCA -3′. In 25 µL di miscela PCR, era composta da DNA genomico 0.5 ng, 2X Master Mix (One PCR) di 100 mM Tris-HCl (pH 9.1), 0.1% TritonTMX-100, 200 mM dNTP, 1.5 mM MgCl2, 0.005 U Taq DNA Polymerase e 0.2 µM forward e reverse primer con regolazione di volume con acqua priva di nucleasi. Il termociclatore PCR (Thermo Scientific Hybaid Px2) è stato programmato come segue: (1) denaturazione iniziale per 2 min a 94 ºC per 1 ciclo; (2) denaturazione a 94 ºC per 45 s; annealing a 54 ºC per 45 s, ed estensione a 72 ºC per 1 min per 32 cicli; (3) estensione finale a 72 ºC per 7 min. I campioni sono stati tenuti a 4 ºC fino a ulteriori analisi. I prodotti di PCR di 16S rRNA (~ 1.500 bp) sono stati rilevati su gel di agarosio allo 0,8%, purificati usando il kit di purificazione dei prodotti di PCR (Vivantis, Malaysia), inviati a First Base Co. Ltd. Ltd. (Malesia) per il sequenziamento del DNA. Le sequenze del gene 16S rRNA sono state poi confrontate con altre disponibili in GenBank utilizzando il programma BLASTN (Basic Local Alignment Search Tools)10. L’albero filogenetico è stato costruito utilizzando il metodo Muscle per l’allineamento delle sequenze e il metodo della massima verosimiglianza utilizzando MEGA X con 1.000 repliche di valori bootstrap11. Tutte le sequenze parziali 16S rRNA dei nostri isolati produttori di amilasi sono state depositate sul database NCBI.
Determinazione dell’attività dell’enzima amilasi
Il metodo ha seguito Bhaskara et al. (2011)12. I tre batteri con il più alto Halo: rapporti di colonia sono stati scelti per la determinazione dell’attività dell’enzima amilasi. Ogni isolato è stato subcoltivato in brodo liquido di amido (g/L): 10.0 amido solubile, 10.0 peptone, 20.0 estratto di lievito, 0.05 KH2PO4, 0.015 MnCl2.4H2O, 0.25 MgSO4.7H2O, 0.05 CaCl2.2H2O, 0.01 FeSO4.7H2O e incubato a 37 ºC, 150 rpm per 1, 2 e 3 giorni. Il surnatante chiaro (enzima amilasi extracellulare grezzo) è stato ottenuto dopo la centrifugazione a 10.000g per 15 minuti a 4 ºC. L’estratto grezzo è stato concentrato utilizzando il concentratore di proteine dell’ultracentrifuga MWCO 10 kDa (Vivaspin, Sartorius, UK).
Il metodo DNS13 è stato utilizzato per determinare l’attività amilasica di ogni isolato batterico ad ogni giorno di induzione enzimatica. Un mL di enzima grezzo è stato mescolato con 1 mL di soluzione di amido all’1% in 1 mL di tampone fosfato di sodio 0,05 M pH 7,0. I campioni sono stati incubati a 37°C per 30 minuti. Dopo l’incubazione, 0,5 mL di soluzione DNS sono stati aggiunti ad ogni campione per fermare la reazione e poi bolliti a 100°C per 5 min a bagnomaria. L’intensità del colore della soluzione è stata osservata misurando la densità ottica (OD) con uno spettrofotometro a 575 nm. La lettura è stata confrontata con una soluzione bianca preparata (senza enzima grezzo). I valori OD dei campioni a T30min sono stati sottratti da quelli dei campioni a T0min poiché il glucosio rimaneva ancora nei campioni a T0min dopo il processo di induzione enzimatica a 1, 2, 3 giorni. Il processo è stato eseguito in triplicato. La concentrazione di glucosio prodotta per ogni soluzione è stata ottenuta dalla curva standard del glucosio. L’attività dell’amilasi è stata calcolata. Un’unità di attività dell’amilasi è definita come la quantità di amilasi necessaria per catalizzare la formazione di zucchero riducente che è uguale a 1 µmol di glucosio per min in condizioni di saggio14.
L’enzima grezzo dell’isolato batterico con la più alta attività è stato scelto per ulteriori lavori.
Per determinare l’attività enzimatica specifica degli isolati selezionati, è stato usato il metodo Folin-Lowry per la stima delle proteine totali15. L’attività enzimatica specifica è stata misurata usando la seguente formula.
Attività specifica (U/mg) = Attività enzimatica (U/ml)/Concentrazione proteica extracellulare (mg/ml)
Php ottimale e temperatura dell’attività enzimatica dell’amilasi
Sono state testate soluzioni di amido solubile (1%) in diversi pH da 3 a 10. Un mL di diverse soluzioni di substrato all’1% è stato aggiunto insieme a 1 mL dei rispettivi tamponi: tampone citrato 0,05 M (pH da 3 a 5), tampone fosfato di sodio 0,05 M (pH 6 e 7), Tris-HCl 0,05 M (pH 8 e 9) e NaOH glicina 0,05 M (pH 10). Un mL di enzima grezzo è stato aggiunto anche a questi tamponi. I campioni sono stati incubati a 37°C per 30 minuti. L’attività specifica dell’amilasi è stata calcolata. Il pH al quale è stata osservata la massima attività è stato annotato. Allo stesso modo, sono state preparate diverse soluzioni di substrato sciogliendo l’1% di amido solubile in soluzioni a pH 7.0. Un mL di 1% di amido solubile è stato aggiunto insieme a 1 mL di tampone di fosfato di sodio 0,05 M (pH 7). Un mL di enzima grezzo è stato aggiunto anche ai tamponi. I campioni sono stati incubati a 20, 30, 40, 50, 60, 70 e 80°C per 30 minuti. L’attività specifica dell’amilasi è stata calcolata. È stata annotata la temperatura alla quale è stata osservata la massima attività. Sia il pH che la temperatura ottimali sono stati usati per determinare l’attività enzimatica amilasica specifica finale.
Analisi statistica
Sono state usate l’Analisi della Varianza a una via (ANOVA) con il test di Duncan Multiple Range su SPSS Statistics Ver. 17.0. I risultati sono stati espressi come mezzi ± SD con differenza statistica quando p < 0,05.
Risultati
Isolamento di batteri produttori di amilasi
In questo studio, 13 isolati amilasi-positivi hanno mostrato zone chiare su agar amido 1% con diversi rapporti Halo : Colonia. Le colonie che mostravano zone chiare di soluzione di iodio sono state prese come colonie batteriche positive per la degradazione dell’amido. Tutti gli isolati batterici hanno mostrato morfologie di colonie simili e sono apparsi come batteri Gram-positivi e a forma di asta (Tabella 1). Il risultato ha mostrato Halo: rapporti di colonia che vanno da 1,18 a 1,71 (Tabella 1). Gli isolati batterici sono stati quindi utilizzati per determinare la durata ottimale di induzione dell’enzima amilasi e solo i 3 isolati con le attività specifiche più elevate sono stati mostrati in 3.4.
Identificazione del ceppo dei batteri produttori di amilasi
Tutti i 13 ceppi batterici amilasi-positivi sono stati sottoposti al sequenziamento del gene 16S rRNA per l’identificazione del ceppo. I risultati BLAST hanno mostrato che tutti gli isolati positivi all’amilasi appartengono al genere Bacillus, tranne un isolato appartenente a Enterobacter (Tabella 2). I loro parenti più prossimi sono stati trovati in India, Cina, Corea, Indonesia, Argentina, Italia, Israele, USA, Argentina e Sudafrica con una gamma di identità del 96-100%.
Analisi filogenetica
L’albero filogenetico di 13 ceppi batterici amilasi-positivi e 2 ceppi di riferimento con enzimi amilasi putativi usando MEGA 7.0 ha mostrato che E. cloacae 2.1AL2 era evolutivamente diverso dagli altri ceppi Bacillus (Fig. 1). Il ceppo di riferimento B. thuringiensis serovar konkukian (NCBI accession no. AB617494.1) si è evoluto diversamente dai nostri batteri Bacillus. Tuttavia, un altro ceppo di riferimento B. cereus ATCC 14579 (NCBI accession no. MG708176.1) isolato dalla foglia di cartamo, Eqypt mostrato evoluzione simile ai nostri batteri (Fig. 1).
 |
Tabella 1: Caratteristiche e Halo : Colony ratios of 13 amylase-producing bacterial strains
Click here to View table |
Optimal amylase enzyme induction duration
It was shown that the incubation duration that induced amylase production with the highest specific activity was 2-3 days among all 3 isolates without statistic differences. Bacillus sp. 3.5AL2 sembrava produrre la più alta attività specifica a 0,91 ± 0,12 U/mg al giorno 2 a 37°C (Tabella 3). Così, questo ceppo e questa condizione sono stati utilizzati per indurre la produzione di amilasi per ulteriori analisi.
Phptimal pH e temperatura dell’attività dell’enzima amilasi
Bacillus sp. 3.5AL2 ha mostrato la più alta attività specifica a pH 7.0 a 37°C e a 60°C a pH 7.0 (Fig. 2). Così, entrambe le condizioni ottimali sono state utilizzate per determinare l’attività specifica dell’amilasi e si è ottenuto 1,97 ± 0,41 U/mg (Tabella 4).
Tabella 2: Tredici ceppi batterici positivi all’amilasi identificati dall’analisi 16S rRNA
| Isolato | Accessione
no. a |
Parente più prossimob | Numero di accessi c | % Identitàd | Origine |
| 1.1AL1 | MK578206 | Bacillus pseudomycoides FJAT-hcl-17 | KY653098.1 | 99% | Host Paris polyphylla var. chinensis, Cina |
| 1.1AL2 | MK578207 | Bacillus sp. 188Cu-As | KM349197.1 | 99% | Biofilm, Argentina |
| 1.2AL3 | MK578208 | Bacillus anthracis SAK4 | MG706137.1 | 100% | Suolo, Repubblica di Corea |
| 1.4AL1 | MK578209 | Bacillus sp. CNJ732 PL04 | DQ448749.1 | 99% | Sedimento marino, USA |
| 1.4AL3 | MK578210 | Bacillus cereus B19 | MK229038.1 | 99% | Grano di grano, Isarael |
| 2.1AL2 | MK578211 | Enterobacter cloacae HNXY160623 | KX431213.1 | 98% | Embrione aviario, Cina |
| 2.3AL1 | MK578212 | Bacillus thuringiensis BD17-E12 | HF584771.1 | 99% | Sistema radicale della vite, Italia |
| 2.3AL8 | MK578213 | Bacillus cereus DFT-1 | KY750685.1 | 98% | Acqua di mare della zona industriale, Indonesia |
| 2.3AL9 | MK578214 | Bacillus sp. PTP1 | KY910137.1 | 96% | Budello di cocciniglia della papaia, India |
| 2.4AL2 | MK578215 | Bacillus pseudomycoides 74 | MH910178.1 | 99% | India |
| 3.2AL1 | MK578216 | Bacillus thuringiensis
IARI-IIWP-38 |
KF054891.1 | 99% | Rizospere di grano, India |
| 3.4AL1 | MK578217 | Bacillus cereus F4a | MK088302.1 | 99% | Suolo della rizosfera del tè, India |
| 3.5AL2 | MK578218 | Bacillus cereus SP1-AB4 | MH013307.1 | 99% | Spugna marina, Sud Africa |
aAdesione GenBank no. dei nostri ceppi depositati sul sito NCBI (http://www.ncbi.nlm.nih.gov/pubmed)
bSpecie più vicine con la più alta % di identità e il più alto punteggio Max sulla ricerca BLAST
cGenBank accession no. dei ceppi parenti più vicini sul sito NCBI
dB Sulla base dei risultati della ricerca BLAST, identità (%) dei ceppi rispetto ai
parenti più vicini.
eBasato sui risultati della ricerca BLAST, origine dei parenti più vicini.
 |
Figura 1: Albero filogenetico di 13 ceppi batterici positivi all’amilasi e 2 ceppi di Bacillus di riferimento
Clicca qui per vedere la figura |
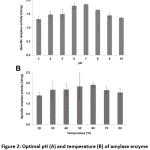 |
Figura 2: pH ottimale (A) e temperatura (B) dell’attività dell’enzima amilasi di Bacillus sp. 3.5AL2
Clicca qui per vedere la figura |
Tabella 3: Durata ottimale dell’induzione dell’enzima amilasi
|
Isolato |
Attività amilasi specifica attività
(U/mg di proteina) |
||
| 24 h | 48 h | 72 h | |
| Bacillus sp. 1.1AL2 | 0,21 ± 0,04Ab | 0,40 ± 0,04Ca | 0,41 ± 0,04Ba |
| B. cereus 2.3AL8 | 0,45 ± 0,06Ab | 0,62 ± 0,04Ba | 0,65 ± 0,06Aa |
| Bacillus sp. 3.5AL2 | 0,32 ± 0,11Ab | 0,91 ± 0,12Aa | 0,86 ± 0,11Aa |
I valori sono medi ± SD in triplicato.
Lettere maiuscole e minuscole indicano differenze statistiche (p < 0,05) nella colonna e nella riga, rispettivamente.
Tabella 4: Attività enzimatica specifica dell’amilasi a pH e temperatura ottimali
| Strain | Activity (U/mg) | Optimal Temp. (°C) | Php ottimale |
| Bacillus sp. 3.5AL2 | 1.97 ± 0.41 | 60 | 7.0 |
Discussione
I dati doganali thailandesi (2017) hanno mostrato che la Thailandia ha importato enzimi industriali da altri paesi con il valore di 2.625 milioni di Bahts nel 2016 e il tasso di importazione è stato aumentato del 9,40% all’anno. Uno degli enzimi più utilizzati in Thailandia, l’amilasi, è stato applicato nell’industria dell’abbigliamento, tessile e alimentare. Tuttavia, c’è una mancanza di produzione locale di amilasi commerciale e quindi questo lavoro mirava a isolare i batteri del suolo dalla foresta locale con la capacità di produrre amilasi per usi industriali.
I residui ricchi di amido possono essere una fonte potenziale migliore dove i batteri positivi all’amilasi possono essere isolati16. Inoltre, questi potrebbero essere isolati da luoghi come le fattorie di manioca, il suolo e le fabbriche di farina di trasformazione17. In questo lavoro, 13 batteri produttori di amilasi sono stati isolati e identificati come Bacillus spp. e Enterobacter sp. che è simile ai rapporti precedenti18, 19, 20 dove Bacillus spp. sono stati trovati principalmente come produttori di amilasi.
I nostri risultati hanno mostrato che Bacillus sp. 3.5AL2, l’isolato con la più alta attività specifica tra 13 isolati, ha prodotto la più alta attività specifica a 0,91 ± 0,12 U/mg a 2 giorni di incubazione a 37°C. Il pH e la temperatura ottimali per l’attività amilasica erano rispettivamente 7,0 e 60°C. Entrambe le condizioni ottimali hanno dato l’attività amilasica specifica di Bacillus sp. 3.5AL2 di 1,97 ± 0,41 U/mg. È noto che gli enzimi che possono lavorare ad alta temperatura sono considerati un vantaggio nei processi industriali come la liquefazione dell’amido21. Quindi, l’amilasi da Bacillus sp. 3.5AL2 ha un potenziale uso in tali industrie.
Similmente, Bacillus sp. GM890 ha mostrato la temperatura ottimale per l’amilasi a 60°C20, Bacillus licheniformis AI20 mostra la massima attività tra l’intervallo di 60-80°C22 e Bacillus sp. WA21 aveva una temperatura ottimale inferiore di 55°C per l’amilasi19.
La maggior parte dei ceppi batterici che degradano l’amido ha rivelato un intervallo di pH compreso tra 6,0 e 7,0 per la crescita normale e l’attività enzimatica4. Allo stesso modo, in questo lavoro, Bacillus sp. 3.5AL2 aveva un pH ottimale a 7.0 che non richiede alcuna aggiunta di acido o alcali. La nostra scoperta era in accordo con Bacillus sp. isolato dai rifiuti dell’industria dhal che mostra una temperatura ottimale di 60°C23 e B. amyloliquefaciens con un pH ottimale di 7.024. Tuttavia, Bacillus sp. WA21 ha mostrato il pH ottimale dell’amilasi a 6.0 che è inferiore a quello trovato in questo studio19.
Questo tipo di studio è molto importante per il punto di partenza della produzione commerciale di amilasi in Thailandia. Tuttavia, ulteriori indagini di ricerca sono essenziali per rendere la produzione di amilasi economicamente vantaggiosa. C’è bisogno di un’accurata caratterizzazione dell’enzima, di un’ulteriore determinazione della termostabilità, della stabilità del pH, dell’influenza di diversi ioni metallici e di diversi substrati, specialmente i rifiuti agricoli in Thailandia, e delle loro concentrazioni.
Dopo aver ottenuto tutti i dati necessari come menzionato sopra, si pensa che la produzione di amilasi commerciale nelle industrie enzimatiche della Thailandia sarà in aumento e diffusa in ogni regione e a sua volta andrà a beneficio dell’economia del paese a causa dell’autosufficienza sulle proprie risorse per produrre l’enzima amilasi.
Conclusione
Questa è la prima relazione di identificazione di 13 isolati batterici produttori di amilasi dal suolo nella foresta comunitaria Nasinuan, Maha Sarakham. Tutti i batteri sono stati identificati come Bacillus spp. tranne uno del genere Enterobacter. Questi batteri possono essere usati per la produzione di amilasi e applicati a livello locale e nazionale nell’agricoltura, nella trasformazione alimentare e nelle industrie tessili in futuro. Così, questo ridurrà il costo dell’importazione di enzimi industriali da altri paesi, offrirà la sostenibilità della produzione locale di enzimi e migliorerà l’economia della nazione.
Riconoscimenti
Gli autori desiderano ringraziare la Mahasarakham University (Grant Year 2018) per il sostegno finanziario assegnato a SD, e la Facoltà di Tecnologia, Mahasarakham University (Grant Year 2017) assegnato a VL.
Conflitto di interessi
Gli autori non dichiarano alcun conflitto di interessi.
- Pandey A. Glucoamylase research: Una panoramica. 47:439-445 (1995).
- Burhan A.I., Nisa U., Gokhan C., Omer C., Ashabil A., and Osman G. Enzymatic properties of a novel thermostable, thermophilic, alkaline and chelator resistant amylase from an alkaliphilic Bacillus isolate ANT-6. Processo Biochem. 38: 1397-1403 (2003).
- Gurung N., Ray S., Bose S., and Rai, V.A. Broader view: Enzimi microbici e la loro rilevanza nelle industrie, nella medicina e oltre. Biomed Res Internat. 1-18. doi:10.1155/2013/ 329121 (2013).
- Gupta R., Gigras P., Mohapatra H., Goswami V.K., e Chauhan B. Microbial α-amilasi: una prospettiva biotecnologica. Process Biochem. 38:1599-1616 (2003).
- Takasaki Y. Un’amilasi che produce maltosio e maltopentosio da circulans. Agric Biol Chem. 47:2193-2199 (1983).
- Fogarty W.M., and Kelly C.T. Amilasi, amiloglucosidasi e relative glucanasi. Rose A.H. Economic Microbiology, Microbial Enzymes and Bioconversion, New York Academic Press Inc, vol. 5. Pp. 115-170 (1980).
- Wind R.D., Buitelaar R.M.G., Huizing H.J., and Dijkhuizen L. Characterization of a new Bacillus stearothermophilus isolate: a highly thermostable α-amylase producing strain. Appl Microbiol Biotechnol. 41:155-162 (1994).
- Brumm P.J., Hebeda R.E., and Teague W.M. Purificazione &caratterizzazione dell’α-amilasi commercializzata e clonata da Megaterium. Parte I: purificazione & proprietà idrolitiche. Amido. 43:319-323 (1991).
- Takasaki Y. Un’amilasi che produce maltotriose da subtilis. Agric Bio. Chem. 49:1091-1097 (1985).
- Lowry O.H., Rosebrough N.J., and Randall R.J. Protein measurement with the folin phenol reagents. J Biol Chem. 193: 265-275 (1951).
- Altschul S.F., Gish W., Miller W., Myers E.W., and Lipman D.J. Basic local alignment search tool. J Mol 215:403-410 (1990).
- Bhaskara R.K.V., Ashwini K., Gaurav K., and Karthik L. Optimization, production and partial purification of extracellular α-amylase from Bacillus marini. Arc Appl Sci Res. 3:33-42 (2011).
- GhoseK. Misura delle attività della cellulasi. Pure Appl Chem. 59: 257-268 (1987).
- Haq U.I., Ashraf H., Iqbal J., and Qadeer M.A. Production of α-amylase by Bacillus licheniformis using an economical medium. Biores Technol. 87:57-61 (2003).
- Mishra S., e Behera N. Attività di amilasi di batteri che degradano l’amido isolati dal suolo che riceve rifiuti di cucina. Afr J Biotechnol. 7: 3326-3331 (2008).
- Fossi B.T., Taveaand F., and Ndjonenkeu T. Production and Partial Characterization of a Themostable amylase from Ascomycetes yeast strain isolated from starchy soils. Afr J Biotechnol. 4:14-18 (2005).
- Cordeiro C.A.M. Production and propertiesof α-amylase from thermophilic Bacillus sp. Brazil J Microbiol. 33:57-61 (2002).
- Asad W. Produzione di enzimi extracellulari da batteri termofili indigeni: Purificazione parziale e caratterizzazione dell’α-amilasi di Bacillus sp. WA21. Pakistan J Botany. 43:1045-1052 (2011).
- Kim T.U. Purificazione e caratterizzazione di una α-amilasi alcalina che forma maltotetraosio da un ceppo di Bacillus alcalofilo, GM8901. Appl Envin Microbiol. 61: 83105-3112 (1995).
- Sivaramakrishnan S., Gangadharan D., Madhavan K.N., and Pandey A. Coltivazione solida di Bacillus amyloliquefaciens per la produzione di alfa amilasi. Food Technol 44:269-274 (2006).
- Abdel-Fattah Y.R., Soliman N.A., El-Toukhy N.M., El-Gendi H., e Ahmed R.S. Purificazione e caratterizzazione di α-amilasi termostabile prodotta da Bacillus licheniformis isolato AI20. J Chem. 2013:1-11 (2013).
- Thippeswamy S., Girigowda K., e Mulimani V.H. Isolamento e identificazione di α-amilasi producendo Bacillus da Dhal
rifiuti industriali. Indian J Biochem Biophysics. 43: 295-298 (2006). - Abd-ElhalemT., El-Sawy M., Rawia F.G., and Khadiga A.A.T. Production of amylase from Bacillus amyloliquefaciens under submerged fermentation using some agro-industrialby-products. Ann Agri Sci. 60:193-202 (2015).
- Tamura K., e Nei M. Stima del numero di sostituzioni nucleotidiche nella regione di controllo del DNA mitocondriale negli esseri umani e scimpanzé. Mol Biol Evol. 10:512-526 (1993).

(Visitato 12.737 volte, 21 visite oggi)