Manuscrit accepté le :29 Août 19
Publié en ligne le : 18-09-2019 Vérification du plagiat : Oui
Révisé par : Farooq Sher
Révision par : Grigorios Kyriakopoulos
Approbation finale par : Ayush Dogra
Comment Citeclose | Historique des publications
Vijitra Luang-In1* , Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 et Kedsukon Maneewan1
, Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 et Kedsukon Maneewan1
1Unité de recherche sur les antioxydants naturels, Département de biotechnologie, Faculté de technologie, Université Mahasarakham, Khamriang, Kantarawichai, Maha Sarakham, 44150, Thaïlande.
2Collège international d’innovation numérique, Université de Chiang Mai, Chiang Mai 50200, Thaïlande.
Correspondant de l’auteur E-mail : [email protected]
DOI : https://dx.doi.org/10.13005/bpj/1735
Abstract
Cette étude visait à isoler et à identifier les bactéries qui peuvent produire une enzyme amylase à partir de la forêt inexplorée de Nasinuan, district de Kantarawichai, province de Mahasarakham, Thaïlande. Treize isolats bactériens ayant une capacité de production d’amylase sur une gélose d’amidon à 1% ont été identifiés en utilisant le séquençage de l’ARNr 16S. Douze bactéries étaient gram-positives, en forme de bâtonnets et identifiées comme Bacillus spp. et une bactérie gram-négative et en forme de bâtonnets était Enterobacter cloacae. Leurs plus proches parents ont été trouvés en Inde, en Chine, en Corée, en Indonésie, en Argentine, en Italie, en Israël, aux Etats-Unis et en Afrique du Sud. Ces bactéries ont été testées pour leur activité amylasique spécifique après 1-3 jours d’induction enzymatique avec 1% d’amidon à 37°C. Les résultats ont montré l’activité spécifique la plus élevée au jour 2 d’incubation, dans l’ordre : Bacillus cereus 3.5AL2 > 3.4AL1 > 1.4AL3 et donc l’induction enzymatique de 2 jours a été choisie pour une analyse plus approfondie. On a constaté que Bacillus sp. 3.5AL2 présentait l’activité enzymatique spécifique de l’amylase la plus élevée, soit 1,97 ± 0,41 U/mg de protéine, dans les conditions optimales de 60°C et de pH 7,0, après 30 minutes d’incubation avec 1 % d’amidon dans un tampon PBS 0,05 M. Cette souche bactérienne productrice d’amylase offre un grand potentiel pour des applications dans les industries alimentaires et agricoles en Thaïlande.
Mots clés
Amylase ; Bactéries ; forêt de Nasinuan ; sol
Télécharger cet article sous :![]()
Copiez ce qui suit pour citer cet article:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation et identification des bactéries productrices d’amylase du sol dans la forêt communautaire de Nasinuan, Maha Sarakham, Thaïlande. Biomed Pharmacol J 2019;12(3).
Copiez ce qui suit pour citer cette URL:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation et identification des bactéries productrices d’amylase du sol dans la forêt communautaire de Nasinuan, Maha Sarakham, Thaïlande. Biomed Pharmacol J 2019;12(3). Disponible à partir de : http://biomedpharmajournal.org/?p=28497
Introduction
Les amylases hydrolytiques de l’amidon (α-amylase, β-amylase et glucoamylase) sont parmi les enzymes les plus utilisées dans la biotechnologie actuelle. Les glucoamylases (1,4-α-D-glucan glucanohydrolase ; EC 3.2.1.3) ou amyloglucosidases, sont des amylases à action exogène qui produisent du glucose à partir de l’extrémité non réductrice de l’amidon et des oligosaccharides correspondants. Les glucoamylases sont mises en œuvre commercialement pour la transformation des malto-oligosaccharides en glucose1. Bien que les amylases soient produites à partir de différentes sources (micro-organismes, plantes et animaux), les amylases provenant de microbes sont les plus adaptées à la production industrielle en raison de la courte période de croissance microbienne, du faible coût de production, du comportement écologique, des problèmes de manipulation moindres pour les travailleurs, de la productivité2 et de la manipulation facile des gènes bactériens3. Les bactéries et les champignons ont tendance à sécréter des amylases à l’extérieur des cellules pour effectuer la digestion extracellulaire de l’amidon en sucres. Une demande industrielle croissante pour les amylases microbiennes a été observée en raison de leur spécificité de réaction, des conditions douces préalables à la réaction et d’une consommation d’énergie moindre que les méthodes chimiques non enzymatiques conventionnelles.
L’application extensive de l’amylase dans l’alimentation, la liquéfaction de l’amidon, la saccharification, la brasserie, les détergents, le papier, le textile et les industries de distillation, a suscité une plus grande attention pour l’augmentation de la production indigène d’α-amylase4. Bacillus est une source bactérienne commune pour la production industrielle d’amylase. Cependant, les différentes souches présentent des conditions de croissance optimales et un profil de production enzymatique différents. Selon les rapports, les souches de Bacillus ont été largement utilisées industriellement pour produire de l’α-amylase, notamment B. amyloliquefaciens, B. subtilis5, B. licheniformis6, B. stearothermophilus7, B. megaterium8 et B. circulans9.
À ce jour, un certain nombre de bactéries productrices d’amylase a été documenté, cependant, aucune étude sur les bactéries productrices d’amylase du sol de la forêt communautaire de Nasinuan, district de Kantharawichai, province de Mahasarakham, Thaïlande, n’a été rapportée. Cette forêt semble être riche en biodiversité microbienne qui peut être utile dans la production d’enzymes industrielles, y compris l’amylase. Il s’agit du premier rapport d’identification de bactéries productrices d’amylase isolées de la forêt de Nasinuan. Ces amylases bactériennes peuvent être appliquées localement à diverses industries, y compris la pâte et le papier, le textile, le bioéthanol, la brasserie, la transformation des aliments, l’alimentation animale et l’agriculture en Thaïlande à l’avenir.
Matériaux et méthodes
Échantillons de sol
Les échantillons de sol ont été collectés au hasard sous la surface du sol 15 cm et conservés dans des sacs en polystyrène de la forêt communautaire de Nasinuan, district de Kantarawichai, province de Mahasarakham, Thaïlande (superficie de 9.6 hectare ; coordonnées de 16,340941, 103,210799).
Isolation des bactéries productrices d’amylase
L’échantillon de sol (10 g) a été mis en suspension dans 90 ml de solution stérile de NaCl à 0,85 %. Les suspensions (100 µL) de dilutions sérielles ont été étalées sur une gélose à l’amidon (g/L) ; 3,0 extrait de bœuf, 10,0 amidon soluble, 15,0 agar pH 7,0 et incubées à 37 ºC pendant 7 jours. Ensuite, les plaques ont été inondées d’iode de Gram. Toutes les colonies avec formation d’une zone claire autour des colonies ont été sous-cultivées dans un bouillon liquide et étalées au moins cinq fois pour obtenir des isolats purs, comme confirmé par la coloration de Gram et l’observation au microscope optique 1000X. Les isolats purs ont été inoculés ponctuellement sur une gélose à l’amidon et incubés à 37°C pendant 7 jours. Les diamètres des zones claires sur les diamètres des colonies ont été mesurés à l’aide d’une règle comme le rapport Halo : Colonie.
Séquençage du gène de l’ARNr 16S et analyse phylogénétique
Les isolats bactériens purs ont été identifiés à l’aide d’ADN génomiques obtenus par la méthode ci-dessus et d’amorces universelles : amorce directe 27F 5′-GAGAGTTTGATYCTGGCTCAG-3′ et amorce inverse 1492R 5’AAGGAGGTGATCCARCCGCA -3′. Dans 25 µL de mélange PCR, il était composé de 0,5 ng d’ADN génomique, 2X Master Mix (One PCR) de 100 mM Tris-HCl (pH 9,1), 0,1% TritonTMX-100, 200 mM dNTP, 1,5 mM MgCl2, 0,005 U Taq DNA Polymerase et 0,2 µM d’amorce directe et inverse avec ajustement du volume avec de l’eau sans nucléase. Le thermocycleur PCR (Thermo Scientific Hybaid Px2) a été programmé comme suit : (1) dénaturation initiale pendant 2 min à 94 ºC pour 1 cycle ; (2) dénaturation à 94 ºC pendant 45 s ; recuit à 54 ºC pendant 45 s, et extension à 72 ºC pendant 1 min pour 32 cycles ; (3) extension finale à 72 ºC pendant 7 min. Les échantillons ont été conservés à 4 ºC jusqu’à l’analyse ultérieure. Les produits PCR des ARNr 16S (~ 1 500 pb) ont été détectés sur un gel d’agarose à 0,8 %, purifiés à l’aide du kit de purification des produits PCR (Vivantis, Malaisie), envoyés à First Base Co. Ltd. (Malaisie) pour le séquençage de l’ADN. Les séquences du gène de l’ARNr 16S ont ensuite été comparées à d’autres disponibles dans GenBank à l’aide du programme BLASTN (Basic Local Alignment Search Tools)10. L’arbre phylogénétique a été construit en utilisant la méthode Muscle pour l’alignement des séquences et la méthode du maximum de vraisemblance en utilisant MEGA X avec 1 000 répétitions de valeurs bootstrap11. Toutes les séquences partielles d’ARNr 16S de nos isolats producteurs d’amylase ont été déposées sur la base de données NCBI.
Détermination de l’activité enzymatique de l’amylase
La méthode a suivi celle de Bhaskara et al. (2011)12. Les trois bactéries présentant les ratios Halo : Colonie les plus élevés ont été choisies pour la détermination de l’activité enzymatique de l’amylase. Chaque isolat a été subculture dans un bouillon liquide d’amidon (g/L) : 10,0 d’amidon soluble, 10,0 de peptone, 20,0 d’extrait de levure, 0,05 de KH2PO4, 0,015 de MnCl2.4H2O, 0,25 de MgSO4.7H2O, 0,05 de CaCl2.2H2O, 0,01 de FeSO4.7H2O et incubé à 37 ºC, 150 rpm pendant 1, 2 et 3 jours. Le surnageant clair (enzyme amylase extracellulaire brute) a été obtenu après centrifugation à 10 000g pendant 15 minutes à 4 ºC. L’extrait brut a été concentré à l’aide d’un concentrateur de protéines ultracentrifuge MWCO 10 kDa (Vivaspin, Sartorius, UK).
La méthode DNS13 a été utilisée pour déterminer l’activité amylasique de chaque isolat bactérien à chaque jour d’induction enzymatique. Un mL d’enzyme brute a été mélangé avec 1 mL de solution d’amidon à 1 % dans 1 mL de tampon phosphate de sodium 0,05 M pH 7,0. Les échantillons ont été incubés à 37°C pendant 30 minutes. Après l’incubation, 0,5 mL de solution DNS a été ajouté à chaque échantillon pour arrêter la réaction, puis a été bouilli à 100°C pendant 5 min au bain-marie. L’intensité de la couleur de la solution a été observée en mesurant la densité optique (DO) à l’aide d’un spectrophotomètre à 575 nm. La lecture a été comparée à une solution vierge préparée (sans enzyme brute). Les valeurs de DO des échantillons à T30min ont été soustraites de celles des échantillons à T0min puisque le glucose restait encore dans les échantillons à T0min après le processus d’induction enzymatique à 1, 2, 3 jours. Le processus a été réalisé en trois exemplaires. La concentration de glucose produite pour chaque solution a été obtenue à partir de la courbe standard de glucose. L’activité de l’amylase a été calculée. Une unité d’activité amylasique est définie comme la quantité d’amylase nécessaire pour catalyser la formation de sucre réducteur qui est égale à 1 µmol de glucose par minute dans les conditions de l’essai14.
L’enzyme brute de l’isolat bactérien ayant l’activité la plus élevée a été choisie pour la suite du travail.
Pour déterminer l’activité enzymatique spécifique des isolats sélectionnés, la méthode de Folin-Lowry pour l’estimation des protéines totales a été utilisée15. L’activité enzymatique spécifique a été mesurée à l’aide de la formule suivante.
Activité spécifique (U/mg) = Activité enzymatique (U/ml)/Concentration de protéines extracellulaires (mg/ml)
Ph optimal et température de l’activité enzymatique de l’amylase
Des solutions d’amidon soluble (1%) à différents pH de 3 à 10 ont été testées. Un mL de différentes solutions de substrat à 1% a été ajouté avec 1 mL des tampons respectifs ; tampon citrate 0,05 M (pH 3 à 5), tampon phosphate de sodium 0,05 M (pH 6 et 7), Tris-HCl 0,05 M (pH 8 et 9) et glycine NaOH 0,05 M (pH 10). Un mL d’enzyme brute a également été ajouté à ces tampons. Les échantillons ont été incubés à 37°C pendant 30 minutes. L’activité spécifique de l’amylase a été calculée. Le pH auquel l’activité la plus élevée a été observée a été noté. De même, différentes solutions de substrat ont été préparées en dissolvant 1% d’amidon soluble dans des solutions à pH 7.0. Un mL d’amidon soluble à 1% a été ajouté avec 1 mL de tampon phosphate de sodium 0.05 M (pH 7). Un mL d’enzyme brute a également été ajouté aux tampons. Les échantillons ont été incubés à 20, 30, 40, 50, 60, 70 et 80°C pendant 30 minutes. L’activité spécifique de l’amylase a été calculée. La température à laquelle l’activité la plus élevée a été observée a été notée. Le pH et la température optimaux ont été utilisés pour déterminer l’activité enzymatique spécifique de l’amylase finale.
Analyse statistique
L’analyse de variance à une voie (ANOVA à une voie) a été utilisée avec le test de Duncan Multiple Range sur SPSS Statistics Ver. 17.0. Les résultats ont été exprimés en tant que moyennes ± SD avec une différence statistique lorsque p < 0,05.
Résultats
Isolation des bactéries productrices d’amylase
Dans cette étude, 13 isolats positifs à l’amylase ont montré des zones claires sur une gélose d’amidon à 1% avec différents rapports Halo : Colonies. Les colonies montrant des zones claires de la solution d’iode ont été prises comme des colonies bactériennes positives dégradant l’amidon. Tous les isolats bactériens ont montré des morphologies de colonies similaires et sont apparus comme des bactéries Gram-positives et en forme de bâtonnets (Tableau 1). Le résultat a montré des ratios Halo : Colonie allant de 1,18 à 1,71 (Tableau 1). Les isolats bactériens ont ensuite été utilisés pour déterminer la durée optimale d’induction de l’enzyme amylase et seuls les 3 isolats présentant les activités spécifiques les plus élevées ont été présentés en 3.4.
Identification des souches de bactéries productrices d’amylase
Les 13 souches bactériennes amylase-positives ont été soumises au séquençage du gène de l’ARNr 16S pour l’identification des souches. Les résultats de BLAST ont montré que tous les isolats positifs à l’amylase appartiennent au genre Bacillus, à l’exception d’un isolat appartenant à Enterobacter (tableau 2). Leurs plus proches parents ont été trouvés en Inde, en Chine, en Corée, en Indonésie, en Argentine, en Italie, en Israël, aux États-Unis et en Afrique du Sud avec une gamme de 96 à 100 % d’identité.
Analyse phylogénétique
L’arbre phylogénétique de 13 souches bactériennes amylase-positives et de 2 souches de référence avec des enzymes amylases putatives en utilisant MEGA 7.0 a montré que E. cloacae 2.1AL2 était différent sur le plan évolutif des autres souches Bacillus (Fig. 1). La souche de référence B. thuringiensis serovar konkukian (NCBI accession no. AB617494.1) a évolué différemment de nos bactéries Bacillus. Cependant, une autre souche de référence B. cereus ATCC 14579 (NCBI accession no. MG708176.1) isolée de la feuille de carthame, Eqypt a montré une évolution similaire à nos bactéries (Fig. 1).
 |
Tableau 1 : Caractéristiques et Halo : Rapports de colonies de 13 souches bactériennes productrices d’amylase
Cliquez ici pour voir le tableau |
Durée optimale d’induction de l’enzyme amylase
Il a été montré que la durée d’incubation qui a induit la production d’amylase avec l’activité spécifique la plus élevée était de 2-3 jours parmi les 3 isolats sans différences statistiques. Bacillus sp. 3.5AL2 a semblé produire l’activité spécifique la plus élevée à 0.91 ± 0.12 U/mg au jour 2 à 37°C (Tableau 3). Ainsi, cette souche et cette condition ont été utilisées pour induire la production d’amylase pour des analyses ultérieures.
Ph optimal et la température de l’activité enzymatique de l’amylase
Bacillus sp. 3.5AL2 a montré l’activité spécifique la plus élevée à pH 7,0 quand 37°C a été fixé et à 60°C quand pH 7,0 a été fixé (Fig. 2). Ainsi, les deux conditions optimales ont été utilisées pour déterminer l’activité spécifique de l’amylase et 1,97 ± 0,41 U/mg (tableau 4) a été obtenu.
Tableau 2 : Treize souches bactériennes positives à l’amylase identifiées par l’analyse de l’ARNr 16S
| Isolat | No d’accession
. a |
Plus proche parentb | No d’accessionc | % Identityd | Origine |
| 1.1AL1 | MK578206 | Bacillus pseudomycoides FJAT-hcl-17 | KY653098.1 | 99% | Host Paris polyphylla var. chinensis, Chine |
| 1.1AL2 | MK578207 | Bacillus sp. 188Cu-As | KM349197.1 | 99% | Biofilms, Argentine |
| 1.2AL3 | MK578208 | Bacillus anthracis SAK4 | MG706137.1 | 100% | Sol, République de Corée |
| 1.4AL1 | MK578209 | Bacillus sp. CNJ732 PL04 | DQ448749.1 | 99% | Sédiment marin, USA |
| 1.4AL3 | MK578210 | Bacillus cereus B19 | MK229038.1 | 99% | Grain de blé, Isarael |
| 2.1AL2 | MK578211 | Enterobacter cloacae HNXY160623 | KX431213.1 | 98% | Embryon d’oiseau, Chine |
| 2.3AL1 | MK578212 | Bacillus thuringiensis BD17-E12 | HF584771.1 | 99% | Système racinaire de la vigne, Italie |
| 2.3AL8 | MK578213 | Bacillus cereus DFT-1 | KY750685.1 | 98% | Eau de mer de la zone industrielle, Indonésie |
| 2.3AL9 | MK578214 | Bacillus sp. PTP1 | KY910137.1 | 96% | Esprit de cochenille de la papaye, Inde |
| 2.4AL2 | MK578215 | Bacillus pseudomycoides 74 | MH910178.1 | 99% | Inde |
| 3.2AL1 | MK578216 | Bacillus thuringiensis
IARI-IIWP-38 |
KF054891.1 | 99% | Rhizospère de blé, Inde |
| 3.4AL1 | MK578217 | Bacillus cereus F4a | MK088302.1 | 99% | Sol de la rhizosphère du thé, Inde |
| 3.5AL2 | MK578218 | Bacillus cereus SP1-AB4 | MH013307.1 | 99% | Eponge marine, Afrique du Sud |
aNo d’accession de la GenBank. de nos souches déposées sur le site NCBI (http://www.ncbi.nlm.nih.gov/pubmed)
bEspèces les plus proches avec le plus haut % d’identité et le plus haut score Max sur la recherche BLAST
cN° d’accessionGenBank des souches les plus proches sur le site NCBI
dSur la base des résultats de la recherche BLAST, identité (%) des souches par rapport aux
plus proches parents.
eSur la base des résultats de la recherche BLAST, origine des plus proches parents.
 |
Figure 1 : Arbre phylogénétique de 13 souches bactériennes amylase-positives et de 2 souches de référence de Bacillus
Cliquez ici pour voir la figure |
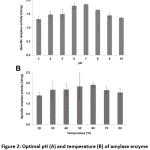 |
Figure 2 : pH optimal (A) et température (B) de l’activité enzymatique de l’amylase de Bacillus sp. 3.5AL2
Cliquez ici pour voir la figure |
Tableau 3 : Durée optimale d’induction de l’enzyme amylase
|
Isolat |
Activité amylasique spécifique activité
(U/mg de protéine) |
||
| 24 h | 48 h | 72 h | |
| Bacillus sp. 1.1AL2 | 0,21 ± 0,04Ab | 0,40 ± 0,04Ca | 0,41 ± 0,04Ba |
| B. cereus 2.3AL8 | 0,45 ± 0,06Ab | 0,62 ± 0,04Ba | 0,65 ± 0,06Aa |
| Bacillus sp. 3.5AL2 | 0,32 ± 0,11Ab | 0,91 ± 0,12Aa | 0,86 ± 0,11Aa |
Les valeurs sont des moyennes ± SD en triplicata.
Les lettres majuscules et minuscules indiquent les différences statistiques (p < 0,05) dans la colonne et la ligne, respectivement.
Tableau 4 : Activité enzymatique spécifique de l’amylase au pH et à la température optimaux
| Souche | Activité (U/mg) | Température optimale. (°C) | Phosphère optimale |
| Bacillus sp. 3.5AL2 | 1,97 ± 0,41 | 60 | 7.0 |
Discussion
Les données des douanes thaïlandaises (2017) ont montré que la Thaïlande a importé des enzymes industrielles d’autres pays avec la valeur de 2,625 millions de bahts en 2016 et le taux d’importation a été augmenté de 9,40% par an. L’une des enzymes les plus utilisées en Thaïlande, l’amylase, a été appliquée dans les vêtements, le textile et les industries alimentaires. Cependant, il y a un manque de production locale de l’amylase commerciale et donc ce travail visait à isoler les bactéries du sol de la forêt locale avec la capacité de produire l’amylase pour les utilisations industrielles.
Résidus riches en amidon peuvent être une meilleure source potentielle où les bactéries amylase-positives peuvent être isolés16. En outre, ceux-ci pourraient être isolés à partir de lieux tels que les fermes de manioc, le sol, et les usines de farine de transformation17. Dans ce travail, 13 bactéries productrices d’amylase ont été isolées et identifiées comme Bacillus spp. et Enterobacter sp. ce qui est similaire aux rapports précédents18, 19, 20 où Bacillus spp. ont été principalement trouvés comme producteurs d’amylase.
Nos résultats ont montré que Bacillus sp. 3.5AL2, l’isolat avec l’activité spécifique la plus élevée parmi 13 isolats, a produit l’activité spécifique la plus élevée à 0,91 ± 0,12 U/mg à 2 jours d’incubation à 37°C. Le pH et la température optimaux pour l’activité amylasique étaient respectivement de 7,0 et 60°C. Ces deux conditions optimales ont donné une activité amylasique spécifique de Bacillus sp. 3.5AL2 de 1,97 ± 0,41 U/mg. Il est connu que les enzymes qui peuvent travailler à haute température sont considérées comme ayant un avantage dans les processus industriels tels que la liquéfaction de l’amidon21. Ainsi, l’amylase de Bacillus sp. 3.5AL2 a une utilisation potentielle dans de telles industries.
De même, Bacillus sp. GM890 a montré la température optimale pour l’amylase à 60°C20, Bacillus licheniformis AI20 montre l’activité la plus élevée entre la gamme de 60-80°C22 et Bacillus sp. WA21 avait une température optimale plus basse de 55°C pour l’amylase19.
La plupart des souches bactériennes dégradant l’amidon ont révélé une gamme de pH entre 6,0 et 7,0 pour une croissance normale et une activité enzymatique4. De même, dans ce travail, Bacillus sp. 3.5AL2 avait un pH optimal à 7,0 qui ne nécessite aucune addition d’acide ou d’alcali. Notre découverte est en accord avec le Bacillus sp. isolé des déchets de l’industrie du dhal qui présente une température optimale de 60°C23 et le B. amyloliquefaciens avec un pH optimal de 7,024. Néanmoins, Bacillus sp. WA21 a montré le pH optimal de l’amylase à 6,0 qui est inférieur à celui trouvé dans cette étude19.
Ce genre d’étude est très important pour le point de départ de la production commerciale d’amylase en Thaïlande. Cependant, des recherches supplémentaires sont essentielles pour rendre la production d’amylase rentable. Il est nécessaire d’effectuer une caractérisation approfondie de l’enzyme, de déterminer davantage la thermostabilité, la stabilité du pH, l’influence de différents ions métalliques et de différents substrats, en particulier les déchets agricoles en Thaïlande, et leurs concentrations.
Après avoir obtenu toutes les données nécessaires comme mentionné ci-dessus, on pense que la production d’amylase commerciale dans les industries enzymatiques de la Thaïlande sera croissante et répandue dans chaque région et à son tour sera bénéfique pour l’économie du pays en raison de l’autonomie sur ses propres ressources pour produire l’enzyme amylase.
Conclusion
C’est le premier rapport d’identification de 13 isolats bactériens producteurs d’amylase à partir du sol dans la forêt communautaire de Nasinuan, Maha Sarakham. Toutes les bactéries ont été identifiées comme des Bacillus spp. sauf une du genre Enterobacter. Ces bactéries peuvent être utilisées pour la production d’amylase et appliquées localement et nationalement dans l’agriculture, la transformation des aliments et les industries textiles à l’avenir. Ainsi, cela réduira le coût de l’importation d’enzymes industrielles d’autres pays, offrira la durabilité de la production locale d’enzymes et améliorera l’économie de la nation.
Remerciements
Les auteurs tiennent à remercier l’Université Mahasarakham (année de subvention 2018) pour le soutien financier accordé à SD, et la Faculté de technologie, Université Mahasarakham (année de subvention 2017) accordée à VL.
Conflit d’intérêt
Les auteurs ne déclarent aucun conflit d’intérêt.
- Pandey A. Recherche sur la glucoamylase : Un aperçu. 47:439-445 (1995).
- Burhan A.I., Nisa U., Gokhan C., Omer C., Ashabil A., et Osman G. Propriétés enzymatiques d’une nouvelle amylase thermostable, thermophile, alcaline et résistante aux chélateurs provenant d’un isolat de Bacillus alcaliphile ANT-6. Process Biochem. 38 : 1397-1403 (2003).
- Gurung N., Ray S., Bose S., et Rai, V.A. Broader view : Les enzymes microbiennes et leur pertinence dans les industries, la médecine et au-delà. Biomed Res Internat. 1-18. doi:10.1155/2013/ 329121 (2013).
- Gupta R., Gigras P., Mohapatra H., Goswami V.K., et Chauhan B. α-amylases microbiennes : une perspective biotechnologique. Process Biochem. 38:1599-1616 (2003).
- Takasaki Y. Une amylase produisant du maltotetrose et du maltopentose à partir de circulans. Agric Biol Chem. 47:2193-2199 (1983).
- Fogarty W.M., et Kelly C.T. Amylase, amyloglucosidase et glucanases apparentées. Rose A.H. Economic Microbiology, Microbial Enzymes and Bioconversion, New York Academic Press Inc, vol. 5. Pp. 115-170 (1980).
- Wind R.D., Buitelaar R.M.G., Huizing H.J., and Dijkhuizen L. Characterization of a new Bacillus stearothermophilus isolate : a highly thermostable α-amylase producing strain. Appl Microbiol Biotechnol. 41:155-162 (1994).
- Brumm P.J., Hebeda R.E., et Teague W.M. Purification &caractérisation de l’α-amylase commercialisée et clonée de megaterium. Partie I : purification & propriétés hydrolytiques. Amidon. 43:319-323 (1991).
- Takasaki Y. Une amylase produisant du maltotriose à partir de subtilis. Agric Bio. Chem. 49:1091-1097 (1985).
- Lowry O.H., Rosebrough N.J., and Randall R.J. Protein measurement with the folin phenol reagents. J Biol Chem. 193 : 265-275 (1951).
- Altschul S.F., Gish W., Miller W., Myers E.W., and Lipman D.J. Basic local alignment search tool. J Mol 215:403-410 (1990).
- Bhaskara R.K.V., Ashwini K., Gaurav K., et Karthik L. Optimisation, production et purification partielle de l’α-amylase extracellulaire de Bacillus marini. Arc Appl Sci Res. 3:33-42 (2011).
- GhoseK. Mesure des activités de la cellulase. Pure Appl Chem. 59 : 257-268 (1987).
- Haq U.I., Ashraf H., Iqbal J., et Qadeer M.A. Production d’α-amylase par Bacillus licheniformis en utilisant un milieu économique. Biores Technol. 87:57-61 (2003).
- Mishra S., et Behera N. L’activité amylasique des bactéries dégradant l’amidon est isolée du sol recevant des déchets de cuisine. Afr J Biotechnol. 7 : 3326-3331 (2008).
- Fossi B.T., Taveaand F., et Ndjonenkeu T. Production et caractérisation partielle d’une amylase Themostable à partir d’une souche de levure Ascomycetes isolée de sols amylacés. Afr J Biotechnol. 4:14-18 (2005).
- Cordeiro C.A.M. Production et propriétésde l’α-amylase de Bacillus sp thermophile. Brazil J Microbiol. 33:57-61 (2002).
- Asad W. Production d’enzymes extracellulaires par des bactéries thermophiles indigènes : Purification partielle et caractérisation de l’α-amylase par Bacillus sp. WA21. Pakistan J Botany. 43:1045-1052 (2011).
- Kim T.U. Purification et caractérisation d’une α-amylase alcaline formant du maltotétraose à partir d’une souche de Bacillus alcalophile, GM8901. Appl Envin Microbiol. 61 : 83105-3112 (1995).
- Sivaramakrishnan S., Gangadharan D., Madhavan K.N., et Pandey A. Culture solide de Bacillus amyloliquefaciens pour la production d’alpha amylase. Food Technol 44:269-274 (2006).
- Abdel-Fattah Y.R., Soliman N.A., El-Toukhy N.M., El-Gendi H., and Ahmed R.S. Purification, and characterization of thermostable α-amylase produced by Bacillus licheniformis isolate AI20. J Chem. 2013:1-11 (2013).
- Thippeswamy S., Girigowda K., et Mulimani V.H. Isolation et identification de Bacillus produisant de l’α-amylase à partir de déchets industriels Dhal
. Indian J Biochem Biophysics. 43 : 295-298 (2006). - Abd-ElhalemT., El-Sawy M., Rawia F.G., et Khadiga A.A.T. Production d’amylase à partir de Bacillus amyloliquefaciens sous fermentation submergée en utilisant certainsby-produits agro-industriels. Ann Agri Sci. 60:193-202 (2015).
- Tamura K., et Nei M. Estimation du nombre de substitutions nucléotidiques dans la région de contrôle de l’ADN mitochondrial chez les humains et les chimpanzés. Mol Biol Evol. 10:512-526 (1993).

(Visité 12 737 fois, 21 visites aujourd’hui).