Manuskript angenommen am :29 Aug 19
Published online on: 18-09-2019 Plagiatsprüfung: Yes
Reviewed by: Farooq Sher
Zweites Review von: Grigorios Kyriakopoulos
Endgültige Freigabe durch: Ayush Dogra
How to Citeclose | Publication Historyclose
Vijitra Luang-In1* , Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 und Kedsukon Maneewan1
, Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 und Kedsukon Maneewan1
1Natural Antioxidant Innovation Research Unit, Department of Biotechnology, Faculty of Technology, Mahasarakham University, Khamriang, Kantarawichai, Maha Sarakham, 44150, Thailand.
2International College of Digital Innovation, Chiang Mai University, Chiang Mai 50200, Thailand.
Korrespondierender Autor E-Mail: [email protected]
DOI : https://dx.doi.org/10.13005/bpj/1735
Abstract
Diese Studie zielte darauf ab, Bakterien zu isolieren und zu identifizieren, die das Enzym Amylase aus dem unerforschten Nasinuan Forest, Kantarawichai District, Mahasarakham Province, Thailand produzieren können. Dreizehn bakterielle Isolate, die auf 1%igem Stärkeagar Amylase produzieren, wurden mittels 16S rRNA-Sequenzierung identifiziert. Zwölf Bakterien waren gram-positiv, stäbchenförmig und wurden als Bacillus spp. identifiziert, ein Bakterium mit gram-negativem und stäbchenförmigem Charakter war Enterobacter cloacae. Ihre nächsten Verwandten wurden in Indien, China, Korea, Indonesien, Argentinien, Italien, Israel, USA, Argentinien und Südafrika gefunden. Diese Bakterien wurden nach 1-3 Tagen Enzyminduktion mit 1% Stärke bei 37°C auf ihre spezifische Amylaseaktivität getestet. Die Ergebnisse zeigten die höchste spezifische Aktivität an Tag 2 der Inkubation in der Reihenfolge: Bacillus cereus 3.5AL2 > 3.4AL1 > 1.4AL3 und daher wurde für die weitere Analyse eine zweitägige Enzyminduktion gewählt. Es wurde festgestellt, dass Bacillus sp. 3.5AL2 die höchste spezifische Amylase-Enzymaktivität von 1,97 ± 0,41 U/mg Protein unter den optimalen Bedingungen von 60°C und pH 7,0 nach 30-minütiger Inkubation mit 1% Stärke in 0,05 M PBS-Puffer aufweist. Dieser Amylase-produzierende Bakterienstamm bietet ein großes Potenzial für Anwendungen in der Lebensmittel- und Agrarindustrie in Thailand.
Schlüsselwörter
Amylase; Bakterien; Nasinuan-Wald; Boden
Diesen Artikel herunterladen als:![]()
Zitieren Sie diesen Artikel wie folgt:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3).
Copy the following to cite this URL:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Isolation and Identification of Amylase-producing Bacteria from Soil in Nasinuan Community Forest, Maha Sarakham, Thailand. Biomed Pharmacol J 2019;12(3). Available from: http://biomedpharmajournal.org/?p=28497
Introduction
Die stärkehydrolytischen Amylasen (α-Amylase, β-Amylase und Glucoamylase) gehören zu den am häufigsten verwendeten Enzymen in der heutigen Biotechnologie. Glucoamylasen (1,4-α-D-Glucan-Glucanohydrolase; EC 3.2.1.3) oder Amyloglucosidasen sind exo-aktive Amylasen, die Glucose aus dem nicht-reduzierenden Ende von Stärke und entsprechenden Oligosacchariden herstellen. Glukoamylasen werden kommerziell für die Umwandlung von Malto-Oligosacchariden in Glukose eingesetzt1. Obwohl Amylasen aus verschiedenen Quellen (Mikroorganismen, Pflanzen und Tiere) hergestellt werden, eignen sich Amylasen aus Mikroben am besten für die industrielle Produktion, da sie eine kurze Wachstumszeit haben, kostengünstig produziert werden können, umweltfreundlich sind, den Arbeitern weniger Probleme bereiten, produktiv sind2 und sich bakterielle Gene leicht manipulieren lassen3. Bakterien und Pilze neigen dazu, Amylasen außerhalb der Zellen abzusondern, um einen extrazellulären Verdau von Stärke zu Zuckern durchzuführen. Aufgrund ihrer Reaktionsspezifität, der milden Reaktionsbedingungen und des geringeren Energieverbrauchs im Vergleich zu den herkömmlichen nicht-enzymatischen chemischen Methoden ist eine steigende industrielle Nachfrage nach mikrobiellen Amylasen zu beobachten.
Die umfangreiche Anwendung von Amylase in der Lebensmittel-, Stärkeverflüssigungs-, Verzuckerungs-, Brauerei-, Waschmittel-, Papier-, Textil- und Destillationsindustrie hat dazu geführt, dass der Steigerung der einheimischen Produktion von α-Amylase größere Aufmerksamkeit geschenkt wird4. Bacillus ist eine gängige bakterielle Quelle für die industrielle Amylaseproduktion. Die verschiedenen Stämme haben jedoch unterschiedliche optimale Wachstumsbedingungen und Enzymproduktionsprofile. Berichten zufolge wurden Bacillus-Stämme in großem Umfang industriell zur Produktion von α-Amylase verwendet, darunter B. amyloliquefaciens, B. subtilis5, B. licheniformis6, B. stearothermophilus7, B. megaterium8 und B. circulans9.
Bislang wurde eine Reihe von Amylase-produzierenden Bakterien dokumentiert, jedoch wurde keine Studie über Amylase-produzierende Bakterien aus dem Boden des Nasinuan Community Forest, Kantharawichai District, Mahasarakham Province, Thailand berichtet. Dieser Wald scheint reich an mikrobieller Biodiversität zu sein, die für die Produktion industrieller Enzyme, einschließlich Amylase, nützlich sein kann. Dies ist der erste Bericht über die Identifizierung von Amylase produzierenden Bakterien, die aus dem Nasinuan-Wald isoliert wurden. Diese bakteriellen Amylasen können in Zukunft in verschiedenen Industriezweigen wie Zellstoff und Papier, Textil, Bioethanol, Brauerei, Lebensmittelverarbeitung, Tierfutter und Landwirtschaft in Thailand eingesetzt werden.
Materialien und Methoden
Bodenproben
Bodenproben wurden nach dem Zufallsprinzip 15 cm unterhalb der Bodenoberfläche entnommen und in Styroporbeuteln aus dem Nasinuan Community Forest, Kantarawichai District, Mahasarakham Province, Thailand (Fläche von 9.6 Hektar; Koordinate von 16.340941, 103.210799).
Isolierung von Amylase-produzierenden Bakterien
Die Bodenprobe (10 g) wurde in 90 mL steriler 0,85%iger NaCl-Lösung suspendiert. Die Suspensionen (100 µL) serieller Verdünnungen wurden auf Stärkeagar (g/L); 3,0 Rindfleischextrakt, 10,0 lösliche Stärke, 15,0 Agar pH 7,0 ausgebracht und bei 37 ºC 7 Tage lang bebrütet. Anschließend wurden die Platten mit Gram’schem Jod geflutet. Alle Kolonien, die eine klare Zone um die Kolonien herum bildeten, wurden in flüssiger Brühe subkultiviert und mindestens fünfmal ausgestrichen, um reine Isolate zu erhalten, was durch Gram-Färbung und 1000-fache lichtmikroskopische Beobachtung bestätigt wurde. Die reinen Isolate wurden punktuell auf Stärkeagar beimpft und 7 Tage lang bei 37°C bebrütet. Die Durchmesser der klaren Zonen über den Durchmessern der Kolonien wurden mit einem Lineal als Halo : Kolonie-Verhältnis gemessen.
16S rRNA-Gen-Sequenzierung und phylogenetische Analyse
Reine bakterielle Isolate wurden mit genomischer DNA, die mit der oben genannten Methode gewonnen wurde, und universellen Primern identifiziert: Vorwärtsprimer 27F 5′-GAGAGTTTGATYCTGGCTCAG-3′ und Rückwärtsprimer 1492R 5’AAGGAGGTGATCCARCCGCA -3′. Die 25 µL PCR-Mischung bestand aus 0,5 ng genomischer DNA, 2X Master Mix (One PCR) aus 100 mM Tris-HCl (pH 9,1), 0,1% TritonTMX-100, 200 mM dNTP, 1,5 mM MgCl2, 0,005 U Taq DNA Polymerase und 0,2 µM Vorwärts- und Rückwärtsprimer, wobei das Volumen mit nukleasefreiem Wasser eingestellt wurde. Der PCR-Thermocycler (Thermo Scientific Hybaid Px2) wurde wie folgt programmiert: (1) anfängliche Denaturierung für 2 Minuten bei 94 ºC für einen Zyklus; (2) Denaturierung bei 94 ºC für 45 Sekunden; Annealing bei 54 ºC für 45 Sekunden und Verlängerung bei 72 ºC für 1 Minute für 32 Zyklen; (3) endgültige Verlängerung bei 72 ºC für 7 Minuten. Die Proben wurden bis zur weiteren Analyse bei 4 ºC aufbewahrt. Die PCR-Produkte der 16S rRNA (~ 1.500 bp) wurden auf einem 0,8%igen Agarosegel nachgewiesen, mit dem PCR-Produktreinigungskit (Vivantis, Malaysia) gereinigt und an First Base Co. Ltd. (Malaysia) zur DNA-Sequenzierung geschickt. Die 16S rRNA-Gensequenzen wurden dann mit anderen in GenBank verfügbaren Sequenzen unter Verwendung des BLASTN-Programms (Basic Local Alignment Search Tools)10 verglichen. Der phylogenetische Baum wurde mit der Muscle-Methode für den Sequenzabgleich und der Maximum-Likelihood-Methode unter Verwendung von MEGA X mit 1.000 Wiederholungen der Bootstrap-Werte erstellt11. Alle 16S rRNA-Teilsequenzen unserer Amylase produzierenden Isolate wurden in der NCBI-Datenbank hinterlegt.
Bestimmung der Amylase-Enzymaktivität
Die Methode folgte Bhaskara et al. (2011)12. Für die Bestimmung der Amylase-Enzymaktivität wurden die drei Bakterien mit den höchsten Halo:Kolonie-Verhältnissen ausgewählt. Jedes Isolat wurde in flüssiger Stärkebrühe (g/L) subkultiviert: 10,0 lösliche Stärke, 10,0 Pepton, 20,0 Hefeextrakt, 0,05 KH2PO4, 0,015 MnCl2.4H2O, 0,25 MgSO4.7H2O, 0,05 CaCl2.2H2O, 0,01 FeSO4.7H2O und bei 37 ºC, 150 rpm für 1, 2 und 3 Tage bebrütet. Der klare Überstand (extrazelluläres Amylase-Rohextrakt) wurde nach 15-minütiger Zentrifugation bei 10.000 g bei 4 ºC gewonnen. Der Rohextrakt wurde mit einem Ultrazentrifugen-Proteinkonzentrator (Vivaspin, Sartorius, UK) mit einem MWCO von 10 kDa aufkonzentriert.
Die DNS-Methode13 wurde verwendet, um die Amylaseaktivität jedes Bakterienisolats an jedem Enzyminduktionstag zu bestimmen. Ein mL des rohen Enzyms wurde mit 1 mL einer 1%igen Stärkelösung in 1 mL 0,05 M Natriumphosphatpuffer pH 7,0 gemischt. Die Proben wurden 30 Minuten lang bei 37°C bebrütet. Nach der Inkubation wurden 0,5 ml DNS-Lösung zu jeder Probe gegeben, um die Reaktion zu stoppen, und dann 5 Minuten lang bei 100 °C im Wasserbad gekocht. Die Farbintensität der Lösung wurde durch Messung der optischen Dichte (OD) mit einem Spektrophotometer bei 575 nm ermittelt. Der Messwert wurde mit einer vorbereiteten Blindlösung (ohne rohes Enzym) verglichen. Die OD-Werte der Proben bei T30min wurden von denen der Proben bei T0min subtrahiert, da in den T0min-Proben nach dem Enzyminduktionsprozess nach 1, 2 und 3 Tagen noch Glukose vorhanden war. Der Prozess wurde in dreifacher Ausführung durchgeführt. Die Konzentration der für jede Lösung produzierten Glukose wurde aus der Glukose-Standardkurve ermittelt. Die Aktivität der Amylase wurde berechnet. Eine Einheit der Amylaseaktivität ist definiert als die Menge an Amylase, die erforderlich ist, um die Bildung von reduzierendem Zucker zu katalysieren, was 1 µmol Glukose pro Minute unter Testbedingungen entspricht14.
Das Rohenzym des Bakterienisolats mit der höchsten Aktivität wurde für die weiteren Arbeiten ausgewählt.
Um die spezifische Enzymaktivität der ausgewählten Isolate zu bestimmen, wurde die Folin-Lowry-Methode für die Gesamtproteinschätzung verwendet15. Die spezifische Enzymaktivität wurde nach folgender Formel gemessen:
Spezifische Aktivität (U/mg) = Enzymaktivität (U/ml)/Extrazelluläre Proteinkonzentration (mg/ml)
Optimaler pH-Wert und Temperatur der Amylase-Enzymaktivität
Lösliche Stärkelösungen (1%) mit verschiedenen pH-Werten von 3 bis 10 wurden getestet. Ein mL verschiedener 1%iger Substratlösungen wurde zusammen mit 1 mL der jeweiligen Puffer zugegeben: 0,05 M Citratpuffer (pH 3 bis 5), 0,05 M Natriumphosphatpuffer (pH 6 und 7), 0,05 M Tris-HCl (pH 8 und 9) und 0,05 M Glycin-NaOH (pH 10). Diesen Puffern wurde ebenfalls ein mL des Rohenzyms zugesetzt. Die Proben wurden 30 Minuten lang bei 37°C bebrütet. Die spezifische Amylaseaktivität wurde berechnet. Der pH-Wert, bei dem die höchste Aktivität beobachtet wurde, wurde notiert. Ebenso wurden verschiedene Substratlösungen durch Auflösen von 1 % löslicher Stärke in Lösungen mit einem pH-Wert von 7,0 hergestellt. 1 mL 1 % lösliche Stärke wurde zusammen mit 1 mL 0,05 M Natriumphosphatpuffer (pH 7) hinzugefügt. Den Puffern wurde ebenfalls ein mL des rohen Enzyms zugesetzt. Die Proben wurden 30 Minuten lang bei 20, 30, 40, 50, 60, 70 und 80 °C inkubiert. Die spezifische Amylaseaktivität wurde berechnet. Die Temperatur, bei der die höchste Aktivität beobachtet wurde, wurde notiert. Sowohl der optimale pH-Wert als auch die optimale Temperatur wurden verwendet, um die endgültige spezifische Amylase-Enzymaktivität zu bestimmen.
Statistische Analyse
Einfache Varianzanalyse (One-way ANOVA) wurde mit dem Duncan Multiple Range’s Test auf SPSS Statistics Ver. 17.0 verwendet. Die Ergebnisse wurden als Mittelwerte ± SD ausgedrückt, mit statistischen Unterschieden, wenn p < 0,05.
Ergebnisse
Isolierung von Amylase-produzierenden Bakterien
In dieser Studie zeigten 13 Amylase-positive Isolate klare Zonen auf 1% Stärke-Agar mit unterschiedlichen Halo : Kolonie-Verhältnissen. Die Kolonien, die klare Zonen auf Iodlösung zeigten, wurden als positive Stärke abbauende Bakterienkolonien betrachtet. Alle Bakterienisolate wiesen eine ähnliche Koloniemorphologie auf und erschienen als Gram-positive und stäbchenförmige Bakterien (Tabelle 1). Das Ergebnis zeigte ein Halo : Kolonie-Verhältnis von 1,18 bis 1,71 (Tabelle 1). Die Bakterienisolate wurden dann verwendet, um die optimale Dauer der Amylase-Enzyminduktion zu bestimmen, und nur die drei Isolate mit den höchsten spezifischen Aktivitäten wurden in 3.4 aufgeführt.
Stammidentifizierung von Amylase produzierenden Bakterien
Alle 13 Amylase-positiven Bakterienstämme wurden zur Stammidentifizierung einer 16S rRNA-Gen-Sequenzierung unterzogen. Die BLAST-Ergebnisse zeigten, dass alle Amylase-positiven Isolate zur Gattung Bacillus gehören, mit Ausnahme eines Isolats, das zu Enterobacter gehört (Tabelle 2). Ihre engsten Verwandten wurden in Indien, China, Korea, Indonesien, Argentinien, Italien, Israel, den USA, Argentinien und Südafrika gefunden, wobei die Identität zwischen 96 und 100 % lag.
Phylogenetische Analyse
Der phylogenetische Baum von 13 Amylase-positiven Bakterienstämmen und 2 Referenzstämmen mit putativen Amylase-Enzymen unter Verwendung von MEGA 7.0 zeigte, dass sich E. cloacae 2.1AL2 evolutionär von den anderen Bacillus-Stämmen unterscheidet (Abb. 1). Der Referenzstamm B. thuringiensis serovar konkukian (NCBI-Zugangsnummer AB617494.1) entwickelte sich anders als unsere Bacillus-Bakterien. Ein anderer Referenzstamm B. cereus ATCC 14579 (NCBI-Zugangsnummer MG708176.1), der aus Distelblättern isoliert wurde, zeigte jedoch eine ähnliche Entwicklung wie unsere Bakterien (Abb. 1).
 |
Tabelle 1: Merkmale und Halo : Kolonieverhältnisse von 13 Amylase-produzierenden Bakterienstämmen
Klicken Sie hier, um die Tabelle zu sehen |
Optimale Dauer der Amylase-Enzyminduktion
Es zeigte sich, dass die Inkubationsdauer, die die Amylase-Produktion mit der höchsten spezifischen Aktivität induzierte, bei allen 3 Isolaten 2-3 Tage betrug, ohne statistische Unterschiede. Bacillus sp. 3.5AL2 schien die höchste spezifische Aktivität mit 0,91 ± 0,12 U/mg an Tag 2 bei 37°C zu produzieren (Tabelle 3). Daher wurde dieser Stamm und diese Bedingung für die Induzierung der Amylaseproduktion für weitere Analysen verwendet.
Optimaler pH-Wert und Temperatur der Amylase-Enzymaktivität
Bacillus sp. 3.5AL2 zeigte die höchste spezifische Aktivität bei pH 7,0, wenn 37°C fixiert wurde, und bei 60°C, wenn pH 7,0 fixiert wurde (Abb. 2). Somit wurden beide optimalen Bedingungen zur Bestimmung der spezifischen Amylaseaktivität verwendet und 1,97 ± 0,41 U/mg (Tabelle 4) erhalten.
Tabelle 2: Dreizehn Amylase-positive Bakterienstämme, die durch 16S rRNA-Analyse identifiziert wurden
| Isolat | Hinterlegungsnummer
. a |
Nächster Verwandterb | Zugangs-Nr.c | % Identityd | Origine |
| 1.1AL1 | MK578206 | Bacillus pseudomycoides FJAT-hcl-17 | KY653098.1 | 99% | Host Paris polyphylla var. chinensis, China |
| 1.1AL2 | MK578207 | Bacillus sp. 188Cu-As | KM349197.1 | 99% | Biofilme, Argentinien |
| 1.2AL3 | MK578208 | Bacillus anthracis SAK4 | MG706137.1 | 100% | Boden, Republik Korea |
| 1.4AL1 | MK578209 | Bacillus sp. CNJ732 PL04 | DQ448749.1 | 99% | Marines Sediment, USA |
| 1.4AL3 | MK578210 | Bacillus cereus B19 | MK229038.1 | 99% | Weizenkorn, Isarael |
| 2.1AL2 | MK578211 | Enterobacter cloacae HNXY160623 | KX431213.1 | 98% | Avian embryo, China |
| 2.3AL1 | MK578212 | Bacillus thuringiensis BD17-E12 | HF584771.1 | 99% | Wurzelsystem der Weinrebe, Italien |
| 2.3AL8 | MK578213 | Bacillus cereus DFT-1 | KY750685.1 | 98% | Meerwasser im Industriegebiet, Indonesien |
| 2.3AL9 | MK578214 | Bacillus sp. PTP1 | KY910137.1 | 96% | Papayamehlkäferdarm, Indien |
| 2.4AL2 | MK578215 | Bacillus pseudomycoides 74 | MH910178.1 | 99% | Indien |
| 3.2AL1 | MK578216 | Bacillus thuringiensis
IARI-IIWP-38 |
KF054891.1 | 99% | Weizen-Rhizosphäre, Indien |
| 3.4AL1 | MK578217 | Bacillus cereus F4a | MK088302.1 | 99% | Tee-Rhizosphärenboden, Indien |
| 3.5AL2 | MK578218 | Bacillus cereus SP1-AB4 | MH013307.1 | 99% | Mariner Schwamm, Südafrika |
aGenBank-Zugangsnummer. unserer Stämme, die auf der NCBI-Website hinterlegt sind (http://www.ncbi.nlm.nih.gov/pubmed)
bNächste Spezies mit höchster %-Identität und höchstem Max-Score bei der BLAST-Suche
cGenBank-Zugangsnummer der am nächsten verwandten Stämme auf der NCBI-Website
Basierend auf den BLAST-Suchergebnissen, Identität (%) der Stämme im Vergleich zu den
nächsten Verwandten.
eBasierend auf den BLAST-Suchergebnissen, Herkunft der engsten Verwandten.
 |
Abbildung 1: Phylogenetischer Baum von 13 Amylase-positiven Bakterienstämmen und 2 Bacillus-Referenzstämmen
Klicken Sie hier, um die Abbildung zu sehen |
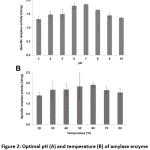 |
Abbildung 2: Optimaler pH-Wert (A) und Temperatur (B) der Amylase-Enzymaktivität von Bacillus sp. 3.5AL2
Click here to View figure |
Tabelle 3: Optimale Amylase-Enzyminduktionsdauer
|
Isolat |
Spezifische Amylase Aktivität
(U/mg Protein) |
||
| 24 h | 48 h | 72 h | |
| Bacillus sp. 1.1AL2 | 0.21 ± 0.04Ab | 0.40 ± 0.04Ca | 0.41 ± 0.04Ba |
| B. cereus 2.3AL8 | 0.45 ± 0.06Ab | 0.62 ± 0.04Ba | 0.65 ± 0.06Aa |
| Bacillus sp. 3.5AL2 | 0,32 ± 0,11Ab | 0,91 ± 0,12Aa | 0,86 ± 0,11Aa |
Werte sind Mittelwerte ± SD in dreifacher Ausführung.
Groß- und Kleinbuchstaben kennzeichnen statistische Unterschiede (p < 0,05) in der Spalte bzw. Zeile.
Tabelle 4: Spezifische Amylase-Enzymaktivität bei optimalem pH-Wert und optimaler Temperatur
| Stamm | Aktivität (U/mg) | Optimal Temp. (°C) | Optimal pH |
| Bacillus sp. 3.5AL2 | 1.97 ± 0.41 | 60 | 7.0 |
Diskussion
Die Daten des thailändischen Zolls (2017) zeigten, dass Thailand im Jahr 2016 Industrieenzyme aus anderen Ländern im Wert von 2.625 Millionen Baht importiert hat und die Importrate um 9,40% pro Jahr gestiegen ist. Eines der am häufigsten verwendeten Enzyme in Thailand, Amylase, wird in der Bekleidungs-, Textil- und Lebensmittelindustrie eingesetzt. Da es jedoch an einer lokalen Produktion von kommerzieller Amylase mangelt, zielte diese Arbeit darauf ab, Bodenbakterien aus lokalen Wäldern zu isolieren, die in der Lage sind, Amylase für industrielle Zwecke zu produzieren.
Stärkereiche Rückstände könnten eine bessere potenzielle Quelle sein, aus der Amylase-positive Bakterien isoliert werden können16. Außerdem könnten sie aus Maniokfarmen, Böden und verarbeitenden Mehlfabriken isoliert werden17. In dieser Arbeit wurden 13 Amylase-produzierende Bakterien isoliert und als Bacillus spp. und Enterobacter sp. identifiziert, was mit früheren Berichten18, 19, 20 übereinstimmt, in denen Bacillus spp. meist als Amylaseproduzenten gefunden wurden.
Unsere Ergebnisse zeigten, dass Bacillus sp. 3.5AL2, das Isolat mit der höchsten spezifischen Aktivität unter den 13 Isolaten, die höchste spezifische Aktivität mit 0,91 ± 0,12 U/mg bei zweitägiger Inkubation bei 37°C produzierte. Der optimale pH-Wert und die optimale Temperatur für die Amylaseaktivität waren 7,0 bzw. 60°C. Bei beiden optimalen Bedingungen lag die spezifische Amylaseaktivität von Bacillus sp. 3.5AL2 bei 1,97 ± 0,41 U/mg. Es ist bekannt, dass Enzyme, die bei hohen Temperaturen arbeiten können, bei industriellen Prozessen wie der Verflüssigung von Stärke von Vorteil sind21. Somit hat die Amylase von Bacillus sp. 3.5AL2 eine potenzielle Verwendung in solchen Industrien.
Auch Bacillus sp. GM890 zeigte die optimale Temperatur für Amylase bei 60°C20, Bacillus licheniformis AI20 zeigte die höchste Aktivität im Bereich von 60-80°C22 und Bacillus sp. WA21 hatte eine niedrigere optimale Temperatur von 55°C für Amylase19.
Die meisten stärkeabbauenden Bakterienstämme zeigten einen pH-Bereich zwischen 6,0 und 7,0 für normales Wachstum und Enzymaktivität4. Auch in dieser Arbeit hatte Bacillus sp. 3.5AL2 einen optimalen pH-Wert von 7,0, der keinen Zusatz von Säure oder Lauge erfordert. Unsere Ergebnisse stimmen mit denen von Bacillus sp. überein, der aus Abfällen der Dhal-Industrie isoliert wurde und eine optimale Temperatur von 60°C23 und B. amyloliquefaciens mit einem optimalen pH-Wert von 7,024 aufweist. Bacillus sp. WA21 zeigte jedoch einen optimalen pH-Wert der Amylase von 6,0, der unter dem in dieser Studie gefundenen Wert liegt19.
Diese Art von Studie ist sehr wichtig für den Beginn der kommerziellen Amylaseproduktion in Thailand. Weitere Forschungsarbeiten sind jedoch unerlässlich, um die Amylaseproduktion kosteneffizient zu gestalten. Es besteht die Notwendigkeit einer gründlichen Enzymcharakterisierung, einer weiteren Bestimmung der Thermostabilität, der pH-Stabilität, des Einflusses verschiedener Metallionen und verschiedener Substrate, insbesondere landwirtschaftlicher Abfälle in Thailand, sowie deren Konzentrationen.
Nach dem Erhalt aller erforderlichen Daten, wie oben erwähnt, wird davon ausgegangen, dass die Produktion von kommerzieller Amylase in der thailändischen Enzymindustrie zunehmen und in jeder Region weit verbreitet sein wird, was wiederum der Wirtschaft des Landes zugute kommen wird, da es auf seine eigenen Ressourcen zur Herstellung von Amylase-Enzym angewiesen ist.
Schlussfolgerung
Dies ist der erste Bericht über die Identifizierung von 13 Amylase-produzierenden bakteriellen Isolaten aus dem Boden im Nasinuan Community Forest, Maha Sarakham. Alle Bakterien wurden als Bacillus spp. identifiziert, außer einem aus der Gattung Enterobacter. Diese Bakterien können für die Amylaseproduktion verwendet und in Zukunft auf lokaler und nationaler Ebene in der Landwirtschaft, der Lebensmittelverarbeitung und der Textilindustrie eingesetzt werden. Dadurch werden die Kosten für den Import von Industrieenzymen aus anderen Ländern gesenkt, die Nachhaltigkeit der lokalen Enzymproduktion gewährleistet und die Wirtschaft des Landes gefördert.
Danksagung
Die Autoren danken der Mahasarakham University (Grant Year 2018) für die finanzielle Unterstützung von SD und der Faculty of Technology, Mahasarakham University (Grant Year 2017) für die Unterstützung von VL.
Interessenkonflikt
Die Autoren erklären, dass kein Interessenkonflikt besteht.
- Pandey A. Glucoamylase research: An overview. 47:439-445 (1995).
- Burhan A.I., Nisa U., Gokhan C., Omer C., Ashabil A., and Osman G. Enzymatic properties of a novel thermostable, thermophilic, alkaline and chelator resistant amylase from an alkaliphilic Bacillus isolate ANT-6. Process Biochem. 38: 1397-1403 (2003).
- Gurung N., Ray S., Bose S., and Rai, V.A. Broader view: Mikrobielle Enzyme und ihre Bedeutung in Industrie, Medizin und darüber hinaus. Biomed Res Internat. 1-18. doi:10.1155/2013/ 329121 (2013).
- Gupta R., Gigras P., Mohapatra H., Goswami V.K., and Chauhan B. Microbial α-amylases: a biotechnological perspective. Process Biochem. 38:1599-1616 (2003).
- Takasaki Y. Eine Amylase zur Produktion von Maltotetrose und von Maltopentose aus Circulans. Agric Biol Chem. 47:2193-2199 (1983).
- Fogarty W.M., and Kelly C.T. Amylase, Amyloglucosidase and related glucanases. Rose A.H. Economic Microbiology, Microbial Enzymes and Bioconversion, New York Academic Press Inc, vol. 5. Pp. 115-170 (1980).
- Wind R.D., Buitelaar R.M.G., Huizing H.J., and Dijkhuizen L. Characterization of a new Bacillus stearothermophilus isolate: a highly thermostable α-amylase producing strain. Appl Microbiol Biotechnol. 41:155-162 (1994).
- Brumm P.J., Hebeda R.E., and Teague W.M. Purification & characterization of commercialized, cloned megaterium α-amylase. Part I: purification & hydrolytic properties. Starch. 43:319-323 (1991).
- Takasaki Y. Eine maltotrioseproduzierende Amylase aus Subtilis. Agric Bio. Chem. 49:1091-1097 (1985).
- Lowry O.H., Rosebrough N.J., and Randall R.J. Protein measurement with the folin phenol reagents. J Biol Chem. 193: 265-275 (1951).
- Altschul S.F., Gish W., Miller W., Myers E.W., and Lipman D.J. Basic local alignment search tool. J Mol 215:403-410 (1990).
- Bhaskara R.K.V., Ashwini K., Gaurav K., and Karthik L. Optimization, production and partial purification of extracellular α-amylase from Bacillus marini. Arc Appl Sci Res. 3:33-42 (2011).
- GhoseK. Measurement of cellulase activities. Pure Appl Chem. 59: 257-268 (1987).
- Haq U.I., Ashraf H., Iqbal J., and Qadeer M.A. Production of α-amylase by Bacillus licheniformis using an economical medium. Biores Technol. 87:57-61 (2003).
- Mishra S., and Behera N. Amylase activity of starch degrading bacteria is isolated from soil receiving kitchen wastes. Afr J Biotechnol. 7: 3326-3331 (2008).
- Fossi B.T., Taveaand F., and Ndjonenkeu T. Production and Partial Characterization of a Themostable amylase from Ascomycetes yeast strain isolated from starchy soils. Afr J Biotechnol. 4:14-18 (2005).
- Cordeiro C.A.M. Production and propertiesof α-amylase from thermophilic Bacillus sp. Brazil J Microbiol. 33:57-61 (2002).
- Asad W. Extracellular enzyme production by indigenous thermophilic bacteria: Partielle Reinigung und Charakterisierung von α-Amylase durch Bacillus sp. WA21. Pakistan J Botany. 43:1045-1052 (2011).
- Kim T.U. Purification and characterization of a maltotetraose-forming alkaline α-amylase from an alkalophilic Bacillus strain, GM8901. Appl Envin Microbiol. 61: 83105-3112 (1995).
- Sivaramakrishnan S., Gangadharan D., Madhavan K.N., and Pandey A. Solid culturing of Bacillus amyloliquefaciens for alpha amylase production. Food Technol 44:269-274 (2006).
- Abdel-Fattah Y.R., Soliman N.A., El-Toukhy N.M., El-Gendi H., and Ahmed R.S. Purification, and characterization of thermostable α-amylase produced by Bacillus licheniformis isolate AI20. J Chem. 2013:1-11 (2013).
- Thippeswamy S., Girigowda K., and Mulimani V.H. Isolation and identification of α-amylase producing Bacillus from Dhal
industry waste. Indian J Biochem Biophysics. 43: 295-298 (2006). - Abd-ElhalemT., El-Sawy M., Rawia F.G., and Khadiga A.A.T. Production of amylase from Bacillus amyloliquefaciens under submerged fermentation using some agro-industrialby-products. Ann Agri Sci. 60:193-202 (2015).
- Tamura K., and Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol Biol Evol. 10:512-526 (1993).

(12.737 mal besucht, 21 Besuche heute)