Manuscrito aceptado el :29-ago-19
Publicado online el: 18-09-2019Comprobación de plagio: Sí
Revisado por: Farooq Sher
Segunda revisión por: Grigorios Kyriakopoulos
Aprobación final por: Ayush Dogra
Cómo citar
Vijitra Luang-In1* , Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 y Kedsukon Maneewan1
, Manatchanok Yotchaisarn1, Worachot Saengha1, Piyachat Udomwong2, Sirirat Deeseenthum1 y Kedsukon Maneewan1
1Unidad de Investigación en Innovación de Antioxidantes Naturales, Departamento de Biotecnología, Facultad de Tecnología, Universidad de Mahasarakham, Khamriang, Kantarawichai, Maha Sarakham, 44150, Tailandia.
2International College of Digital Innovation, Chiang Mai University, Chiang Mai 50200, Tailandia.
Autor correspondiente Correo electrónico: [email protected]
DOI : https://dx.doi.org/10.13005/bpj/1735
Abstract
Este estudio tenía como objetivo aislar e identificar las bacterias que pueden producir la enzima amilasa del inexplorado bosque de Nasinuan, distrito de Kantarawichai, provincia de Mahasarakham, Tailandia. Se identificaron trece aislamientos bacterianos con capacidad de producir amilasa en agar almidón al 1% mediante la secuenciación del ARNr 16S. Doce bacterias eran grampositivas, con forma de bastón y se identificaron como Bacillus spp. y una bacteria con carácter gramnegativo y con forma de bastón era Enterobacter cloacae. Sus parientes más cercanos se encontraron en India, China, Corea, Indonesia, Argentina, Italia, Israel, Estados Unidos y Sudáfrica. Estas bacterias fueron sometidas a pruebas de actividad específica de amilasa después de 1 a 3 días de inducción enzimática con 1% de almidón a 37°C. Los resultados mostraron la mayor actividad específica al segundo día de incubación en el orden siguiente Bacillus cereus 3.5AL2 > 3.4AL1 > 1.4AL3 y, por tanto, se eligió la inducción enzimática de 2 días para el análisis posterior. Se descubrió que Bacillus sp. 3.5AL2 presentaba la mayor actividad enzimática específica de amilasa de 1,97 ± 0,41 U/mg de proteína en las condiciones óptimas de 60°C y pH 7,0 tras 30 minutos de incubación con 1% de almidón en tampón PBS 0,05 M. Esta cepa bacteriana productora de amilasa ofrece un gran potencial para aplicaciones en la industria alimentaria y agrícola en Tailandia.
Palabras clave
Amilasa; Bacterias; Bosque de Nasinuan; suelo
Descargar este artículo como:![]()
Copie lo siguiente para citar este artículo:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Aislamiento e identificación de bacterias productoras de amilasa del suelo en el bosque comunitario de Nasinuan, Maha Sarakham, Tailandia. Biomed Pharmacol J 2019;12(3).
Copie lo siguiente para citar esta URL:
Luang-In V, Yotchaisarn M, Saengha W, Udomwong P, Deeseenthum S, Maneewan K. Aislamiento e identificación de bacterias productoras de amilasa del suelo en el bosque comunitario de Nasinuan, Maha Sarakham, Tailandia. Biomed Pharmacol J 2019;12(3). Disponible en: http://biomedpharmajournal.org/?p=28497
Introducción
Las amilasas hidrolíticas del almidón (α-amilasa, β-amilasa y glucoamilasa) son unas de las enzimas más utilizadas en la biotecnología actual. Las glucoamilasas (1,4-α-D-glucano glucanohidrolasa; EC 3.2.1.3) o amiloglucosidasas, son amilasas de acción exógena que producen glucosa a partir del extremo no reductor del almidón y los oligosacáridos correspondientes. Las glucoamilasas se aplican comercialmente para la transformación de malto-oligosacáridos en glucosa1. Aunque las amilasas se producen a partir de diferentes fuentes (microorganismos, plantas y animales), las amilasas procedentes de microbios son las más adecuadas para la producción industrial debido al corto periodo de crecimiento microbiano, al bajo coste de producción, al comportamiento ecológico, a los menores problemas de manipulación para los trabajadores, a la productividad2 y a la fácil manipulación de los genes bacterianos3. Las bacterias y los hongos tienden a secretar amilasas fuera de las células para realizar la digestión extracelular del almidón en azúcares. Se ha observado un aumento de la demanda industrial de amilasas microbianas debido a su especificidad de reacción, a las condiciones suaves que se requieren para la reacción y al menor consumo de energía que los métodos químicos no enzimáticos convencionales.
La amplia aplicación de la amilasa en las industrias alimentaria, de licuefacción del almidón, de sacarificación, cervecera, de detergentes, papelera, textil y de destilación, ha hecho que se preste mayor atención al aumento de la producción autóctona de α-amilasa4. El Bacillus es una fuente bacteriana común para la producción industrial de amilasa. Sin embargo, diferentes cepas tienen diferentes condiciones óptimas de crecimiento y perfil de producción enzimática. Según los informes, las cepas de Bacillus se han utilizado ampliamente en la industria para producir α-amilasa, incluyendo B. amyloliquefaciens, B. subtilis5, B. licheniformis6, B. stearothermophilus7, B. megaterium8 y B. circulans9.
Hasta la fecha, se ha documentado un número de bacterias productoras de amilasa, sin embargo, no se ha informado de ningún estudio sobre las bacterias productoras de amilasa del suelo del bosque comunitario de Nasinuan, distrito de Kantharawichai, provincia de Mahasarakham, Tailandia. Este bosque parece ser rico en biodiversidad microbiana que puede ser útil para la producción de enzimas industriales, incluida la amilasa. Este es el primer informe que identifica las bacterias productoras de amilasa aisladas del bosque de Nasinuan. Estas amilasas bacterianas pueden aplicarse localmente a varias industrias, incluyendo la de la pulpa y el papel, la textil, la del bioetanol, la cervecera, la del procesamiento de alimentos, la de la alimentación animal y la agrícola en Tailandia en el futuro.
Materiales y Métodos
Muestras de suelo
Las muestras de suelo se recogieron aleatoriamente por debajo de la superficie del suelo 15 cm y se guardaron en bolsas de poliestireno de Nasinuan Community Forest, Kantarawichai District, Mahasarakham Province, Tailandia (área de 9.6 hectáreas; coordenada de 16.340941, 103.210799).
Aislamiento de bacterias productoras de amilasa
La muestra de suelo (10 g) se suspendió en 90 mL de solución estéril de NaCl al 0,85%. Las suspensiones (100 µL) de diluciones seriadas se extendieron en agar almidón (g/L); 3,0 extracto de carne, 10,0 almidón soluble, 15,0 agar pH 7,0 y se incubaron a 37 ºC durante 7 días. A continuación, se inundaron las placas con yodo de Gram. Todas las colonias con formación de una zona clara alrededor de las colonias se subcultivaron en caldo líquido y se sometieron a un proceso de estratificación al menos cinco veces para obtener aislados puros, tal como se confirmó mediante la tinción de Gram y la observación al microscopio de luz de 1000X. Los aislados puros se inocularon por puntos en agar almidón y se incubaron a 37°C durante 7 días. Los diámetros de las zonas claras sobre los diámetros de las colonias se midieron con una regla como relación Halo : Colonia.
Secuenciación del gen del ARNr 16S y análisis filogenético
Se identificaron los aislados bacterianos puros utilizando ADN genómico obtenido con el método anterior y cebadores universales: cebador directo 27F 5′-GAGAGTTTGATYCTGCTCAG-3′ y cebador inverso 1492R 5’AAGGAGGTGATCCARCCGCA -3′. En 25 µL de mezcla de PCR, se compuso de ADN genómico 0,5 ng, 2X Master Mix (One PCR) de 100 mM Tris-HCl (pH 9,1), 0,1% TritonTMX-100, 200 mM de dNTP, 1,5 mM de MgCl2, 0,005 U de Taq ADN Polimerasa y 0,2 µM de cebador directo e inverso con ajuste de volumen con agua libre de nucleasas. El termociclador de PCR (Thermo Scientific Hybaid Px2) se programó como sigue (1) desnaturalización inicial durante 2 min a 94 ºC durante 1 ciclo; (2) desnaturalización a 94 ºC durante 45 s; recocido a 54 ºC durante 45 s, y extensión a 72 ºC durante 1 min durante 32 ciclos; (3) extensión final a 72 ºC durante 7 min. Las muestras se mantuvieron a 4 ºC hasta su posterior análisis. Los productos de la PCR del ARNr 16S (~ 1.500 pb) se detectaron en un gel de agarosa al 0,8%, se purificaron utilizando el kit de purificación del producto de la PCR (Vivantis, Malasia) y se enviaron a First Base Co. Ltd. (Malasia) para la secuenciación del ADN. Las secuencias del gen 16S rRNA se compararon entonces con otras disponibles en GenBank utilizando el programa BLASTN (Basic Local Alignment Search Tools)10. El árbol filogenético se construyó utilizando el método de Muscle para la alineación de secuencias y el método de máxima verosimilitud utilizando MEGA X con 1.000 réplicas de valores bootstrap11. Todas las secuencias parciales del ARNr 16S de nuestros aislados productores de amilasa se depositaron en la base de datos del NCBI.
Determinación de la actividad enzimática de la amilasa
El método siguió a Bhaskara et al. (2011)12. Para la determinación de la actividad enzimática de la amilasa se eligieron las tres bacterias con los mayores ratios Halo: Colonia. Cada aislado se subcultivó en caldo líquido de almidón (g/L): 10,0 de almidón soluble, 10,0 de peptona, 20,0 de extracto de levadura, 0,05 de KH2PO4, 0,015 de MnCl2,4H2O, 0,25 de MgSO4,7H2O, 0,05 de CaCl2,2H2O, 0,01 de FeSO4,7H2O y se incubó a 37 ºC, 150 rpm durante 1, 2 y 3 días. El sobrenadante claro (enzima amilasa extracelular cruda) se obtuvo después de centrifugar a 10.000g durante 15 minutos a 4 ºC. El extracto crudo se concentró utilizando un concentrador de proteínas de ultracentrífuga de MWCO 10 kDa (Vivaspin, Sartorius, Reino Unido).
Se utilizó el método DNS13 para determinar la actividad amilásica de cada aislado bacteriano en cada uno de los días de inducción enzimática. Se mezcló un mL de enzima cruda con 1 mL de solución de almidón al 1% en 1 mL de tampón de fosfato sódico 0,05 M de pH 7,0. Las muestras se incubaron a 37°C durante 30 minutos. Después de la incubación, se añadieron 0,5 mL de solución de DNS a cada muestra para detener la reacción y luego se hirvieron a 100°C durante 5 min en el baño de agua. La intensidad del color de la solución se observó midiendo la densidad óptica (DO) con un espectrofotómetro a 575 nm. La lectura se comparó con una solución en blanco preparada (sin enzima cruda). Los valores de DO de las muestras a T30min se restaron de los de las muestras a T0min, ya que la glucosa aún permanecía en las muestras a T0min después del proceso de inducción enzimática a 1, 2 y 3 días. El proceso se llevó a cabo por triplicado. La concentración de glucosa producida para cada solución se obtuvo a partir de la curva estándar de glucosa. Se calculó la actividad de la amilasa. Una unidad de actividad de amilasa se define como la cantidad de amilasa necesaria para catalizar la formación de azúcares reductores que equivale a 1 µmol de glucosa por minuto en las condiciones del ensayo14.
La enzima bruta del aislado bacteriano que tenía la mayor actividad se eligió para el trabajo posterior.
Para determinar la actividad enzimática específica de los aislados seleccionados, se utilizó el método de Folin-Lowry para la estimación de proteínas totales15. La actividad enzimática específica se midió mediante la siguiente fórmula.
Actividad específica (U/mg) = Actividad enzimática (U/ml)/Concentración de proteína extracelular (mg/ml)
Precisión de pH y temperatura óptimos para la actividad enzimática de la amilasa
Se ensayaron soluciones de almidón soluble (1%) en diferentes pH de 3 a 10. Se añadió un mL de diferentes soluciones de sustrato al 1% junto con 1 mL de los respectivos tampones; tampón citrato 0,05 M (pH 3 a 5), tampón fosfato sódico 0,05 M (pH 6 y 7), Tris-HCl 0,05 M (pH 8 y 9) y NaOH glicina 0,05 M (pH 10). También se añadió un mL de enzima cruda a estos tampones. Las muestras se incubaron a 37°C durante 30 minutos. Se calculó la actividad específica de la amilasa. Se anotó el pH al que se observó la mayor actividad. Asimismo, se hicieron diferentes soluciones de sustrato disolviendo almidón soluble al 1% en soluciones de pH 7,0. Se añadió un mL de almidón soluble al 1% junto con 1 mL de tampón fosfato sódico 0,05 M (pH 7). También se añadió un mL de enzima cruda a los tampones. Las muestras se incubaron a 20, 30, 40, 50, 60, 70 y 80°C durante 30 minutos. Se calculó la actividad específica de la amilasa. Se anotó la temperatura a la que se observó la mayor actividad. Se utilizaron tanto el pH como la temperatura óptimos para determinar la actividad enzimática final de la amilasa específica.
Análisis estadístico
Se utilizó un análisis de varianza de una vía (ANOVA de una vía) con la prueba de rango múltiple de Duncan en SPSS Statistics Ver. 17.0. Los resultados se expresaron como medias ± SD con diferencia estadística cuando p < 0,05.
Resultados
Aislamiento de bacterias productoras de amilasa
En este estudio, 13 aislados positivos a la amilasa mostraron zonas claras en agar almidón al 1% con diferentes relaciones Halo : Colonia. Las colonias que mostraban zonas claras de solución de yodo se tomaron como colonias bacterianas positivas a la degradación del almidón. Todos los aislados bacterianos mostraron morfologías de colonias similares y aparecieron como bacterias Gram-positivas y con forma de bastón (Tabla 1). El resultado mostró ratios Halo : Colonia que iban de 1,18 a 1,71 (Tabla 1). A continuación, se utilizaron los aislamientos bacterianos para determinar la duración óptima de la inducción de la enzima amilasa y sólo se mostraron los 3 aislamientos con mayores actividades específicas en 3.4.
Identificación de las cepas de bacterias productoras de amilasa
Las 13 cepas bacterianas positivas a la amilasa se sometieron a la secuenciación del gen 16S rRNA para la identificación de la cepa. Los resultados del BLAST mostraron que todas las cepas positivas a la amilasa pertenecen al género Bacillus, excepto una cepa que pertenece a Enterobacter (Tabla 2). Sus parientes más cercanos se encontraron en India, China, Corea, Indonesia, Argentina, Italia, Israel, EE.UU. y Sudáfrica con un rango de identidad del 96-100%.
Análisis filogenético
El árbol filogenético de 13 cepas bacterianas positivas a la amilasa y 2 cepas de referencia con enzimas putativas de amilasa utilizando MEGA 7.0 mostró que E. cloacae 2.1AL2 era evolutivamente diferente de las otras cepas de Bacillus (Fig. 1). La cepa de referencia B. thuringiensis serovar konkukian (NCBI accession no. AB617494.1) evolucionó de forma diferente a nuestras bacterias Bacillus. Sin embargo, otra cepa de referencia B. cereus ATCC 14579 (NCBI accession no. MG708176.1) aislada de la hoja de cártamo, Eqypt mostró una evolución similar a la de nuestras bacterias (Fig. 1).
 |
Tabla 1: Características y relaciones Halo : Proporciones de colonias de 13 cepas bacterianas productoras de amilasa
Haga clic aquí para ver la tabla |
Duración óptima de la inducción de la enzima amilasa
Se demostró que la duración de la incubación que indujo la producción de amilasa con la mayor actividad específica fue de 2 a 3 días entre los 3 aislados sin diferencias estadísticas. Bacillus sp. 3.5AL2 pareció producir la mayor actividad específica con 0,91 ± 0,12 U/mg al día 2 a 37°C (Tabla 3). Por lo tanto, se utilizó esta cepa y esta condición para inducir la producción de amilasa para los análisis posteriores.
Preparación óptima de la actividad enzimática de la amilasa
Bacillus sp. 3.5AL2 mostró la mayor actividad específica a pH 7,0 cuando se fijó a 37°C y a 60°C cuando se fijó a pH 7,0 (Fig. 2). Así pues, se utilizaron ambas condiciones óptimas para determinar la actividad amilásica específica y se obtuvo 1,97 ± 0,41 U/mg (Tabla 4).
Tabla 2: Trece cepas bacterianas positivas a la amilasa identificadas mediante el análisis del ARNr 16S
| Aislado | Número de accesión
. a |
Pariente más cercanob | Número de accesoc | % Identidadd | Origen |
| 1.1AL1 | MK578206 | Bacillus pseudomycoides FJAT-hcl-17 | KY653098.1 | 99% | Host Paris polyphylla var. chinensis, China |
| 1.1AL2 | MK578207 | Bacillus sp. 188Cu-As | KM349197.1 | 99% | Biofilms, Argentina |
| 1.2AL3 | MK578208 | Bacillus anthracis SAK4 | MG706137.1 | 100% | Suelo, República de Corea |
| 1.4AL1 | MK578209 | Bacillus sp. CNJ732 PL04 | DQ448749.1 | 99% | Sedimento marino, Estados Unidos |
| 1.4AL3 | MK578210 | Bacillus cereus B19 | MK229038.1 | 99% | Grano de trigo, Isarael |
| 2.1AL2 | MK578211 | Enterobacter cloacae HNXY160623 | KX431213.1 | 98% | Embrión de ave, China |
| 2.3AL1 | MK578212 | Bacillus thuringiensis BD17-E12 | HF584771.1 | 99% | Sistema radicular de la vid, Italia |
| 2.3AL8 | MK578213 | Bacillus cereus DFT-1 | KY750685.1 | 98% | Agua de mar de la zona industrial, Indonesia |
| 2.3AL9 | MK578214 | Bacillus sp. PTP1 | KY910137.1 | 96% | Intestino de cochinilla de la papaya, India |
| 2.4AL2 | MK578215 | Bacillus pseudomycoides 74 | MH910178.1 | 99% | India |
| 3.2AL1 | MK578216 | Bacillus thuringiensis
IARI-IIWP-38 |
KF054891.1 | 99% | Rizoma de trigo, India |
| 3.4AL1 | MK578217 | Bacillus cereus F4a | MK088302.1 | 99% | Suelo de la rizosfera del té, India |
| 3.5AL2 | MK578218 | Bacillus cereus SP1-AB4 | MH013307.1 | 99% | Esponja marina, Sudáfrica |
aGenBank accession no. de nuestras cepas depositadas en el sitio web del NCBI (http://www.ncbi.nlm.nih.gov/pubmed)
bEspecies más cercanas con el mayor % de identidad y la mayor puntuación Max en la búsqueda de BLAST
número de acceso al GenBank de las cepas parientes más cercanas en el sitio web del NCBI
dBasado en los resultados de la búsqueda de BLAST, identidad (%) de las cepas en comparación con los
parientes más cercanos.
eBasado en los resultados de la búsqueda BLAST, origen de los parientes más cercanos.
 |
Figura 1: Árbol filogenético de 13 cepas bacterianas positivas a la amilasa y 2 cepas de referencia de Bacillus
Haga clic aquí para ver la figura |
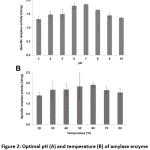 |
Figura 2: pH óptimo (A) y temperatura (B) de la actividad de la enzima amilasa de Bacillus sp. 3.5AL2
Haga clic aquí para ver la figura |
Tabla 3: Duración óptima de la inducción de la enzima amilasa
|
Asolado |
Amilasa específica actividad
(U/mg de proteína) |
||
| 24 h | 48 h | 72 h | |
| Bacillus sp. 1.1AL2 | 0,21 ± 0,04Ab | 0,40 ± 0,04Ca | 0,41 ± 0,04Ba |
| B. cereus 2.3AL8 | 0,45 ± 0,06Ab | 0,62 ± 0,04Ba | 0,65 ± 0,06Aa |
| Bacillus sp. 3.5AL2 | 0,32 ± 0,11Ab | 0,91 ± 0,12Aa | 0,86 ± 0,11Aa |
Los valores son medias ± SD en triplicado.
Las letras mayúsculas y minúsculas indican diferencias estadísticas (p < 0,05) en la columna y en la fila, respectivamente.
Tabla 4: Actividad de la enzima amilasa específica a pH y temperatura óptimos
| Estrella | Actividad (U/mg) | Temperatura óptima. (°C) | PHH óptimo |
| Bacillus sp. 3.5AL2 | 1,97 ± 0,41 | 60 | 7.0 |
Discusión
Los datos de las aduanas tailandesas (2017) mostraron que Tailandia ha importado enzimas industriales de otros países con el valor de 2.625 millones de bahts en 2016 y la tasa de importación se incrementó en un 9,40% por año. Una de las enzimas más utilizadas en Tailandia, la amilasa, se ha aplicado en las industrias de la confección, textil y alimentaria. Sin embargo, hay una falta de producción local de amilasa comercial y, por lo tanto, este trabajo tuvo como objetivo aislar las bacterias del suelo del bosque local con la capacidad de producir amilasa para usos industriales.
Los residuos ricos en almidón pueden ser una mejor fuente potencial donde se pueden aislar las bacterias positivas a la amilasa16. Además, éstas podrían aislarse de lugares como las granjas de yuca, el suelo y las fábricas de harina procesada17. En este trabajo, se aislaron 13 bacterias productoras de amilasa y se identificaron como Bacillus spp. y Enterobacter sp., lo que es similar a los informes anteriores18, 19, 20, en los que se encontraron principalmente Bacillus spp. como productores de amilasa.
Nuestros resultados mostraron que Bacillus sp. 3.5AL2, el aislado con la mayor actividad específica entre los 13 aislados, produjo la mayor actividad específica a 0,91 ± 0,12 U/mg a 2 días de incubación a 37°C. El pH y la temperatura óptimos para la actividad de la amilasa fueron 7,0 y 60°C, respectivamente. Ambas condiciones óptimas dieron una actividad de amilasa específica de Bacillus sp. 3.5AL2 de 1,97 ± 0,41 U/mg. Se sabe que las enzimas que pueden trabajar a alta temperatura se consideran ventajosas en procesos industriales como la licuefacción del almidón21. Así, la amilasa de Bacillus sp. 3.5AL2 tiene un uso potencial en tales industrias.
De manera similar, Bacillus sp. GM890 mostró la temperatura óptima para la amilasa a 60°C20, Bacillus licheniformis AI20 mostró la actividad más alta entre el rango de 60-80°C22 y Bacillus sp. WA21 tuvo una temperatura óptima más baja de 55°C para la amilasa19.
La mayoría de las cepas bacterianas degradadoras de almidón revelaron un rango de pH entre 6,0 y 7,0 para el crecimiento normal y la actividad enzimática4. Asimismo, en este trabajo, Bacillus sp. 3.5AL2 tuvo un pH óptimo de 7,0 que no requiere ninguna adición de ácido o álcali. Nuestro hallazgo concuerda con el de Bacillus sp. aislado de residuos de la industria del dhal, que presenta una temperatura óptima de 60°C23 , y el de B. amyloliquefaciens, con un pH óptimo de 7,024. Sin embargo, Bacillus sp. WA21 mostró el pH óptimo de la amilasa a 6,0 que es menor que el encontrado en este estudio19.
Este tipo de estudio es muy importante para el punto de partida de la producción comercial de amilasa en Tailandia. Sin embargo, es esencial realizar investigaciones adicionales para que la producción de amilasa sea rentable. Es necesario caracterizar a fondo la enzima, determinar más la termoestabilidad, la estabilidad del pH, la influencia de diferentes iones metálicos y diferentes sustratos, especialmente los residuos agrícolas en Tailandia, y sus concentraciones.
Después de obtener todos los datos necesarios como se ha mencionado anteriormente, se cree que la producción de amilasa comercial en las industrias de enzimas de Tailandia será cada vez mayor y generalizada en todas las regiones y, a su vez, será beneficiosa para la economía del país debido a la autosuficiencia de sus propios recursos para producir la enzima amilasa.
Conclusión
Este es el primer informe de la identificación de 13 aislados bacterianos productores de amilasa del suelo en el Bosque Comunitario de Nasinuan, Maha Sarakham. Todas las bacterias fueron identificadas como Bacillus spp. excepto una del género Enterobacter. Estas bacterias pueden utilizarse para la producción de amilasa y aplicarse a nivel local y nacional en la agricultura, el procesamiento de alimentos y las industrias textiles en el futuro. Así, esto reducirá el coste de la importación de enzimas industriales de otros países, ofrecerá la sostenibilidad de la producción local de enzimas y mejorará la economía de la nación.
Agradecimientos
Los autores desean agradecer a la Universidad de Mahasarakham (Año de subvención 2018) por el apoyo financiero concedido a SD, y a la Facultad de Tecnología de la Universidad de Mahasarakham (Año de subvención 2017) concedido a VL.
Conflicto de intereses
Los autores declaran no tener ningún conflicto de intereses.
- Pandey A. Glucoamylase research: Una visión general. 47:439-445 (1995).
- Burhan A.I., Nisa U., Gokhan C., Omer C., Ashabil A., and Osman G. Enzymatic properties of a novel thermostable, thermophilic, alkaline and chelator resistant amylase from an alkaliphilic Bacillus isolate ANT-6. Process Biochem. 38: 1397-1403 (2003).
- Gurung N., Ray S., Bose S., y Rai, V.A. Broader view: Microbial enzymes and their relevance in industries, medicine and beyond. Biomed Res Internat. 1-18. doi:10.1155/2013/ 329121 (2013).
- Gupta R., Gigras P., Mohapatra H., Goswami V.K., y Chauhan B. Microbial α-amylases: a biotechnological perspective. Process Biochem. 38:1599-1616 (2003).
- Takasaki Y. Una amilasa productora de maltotetrosa y de maltopentosa de circulans. Agric Biol Chem. 47:2193-2199 (1983).
- Fogarty W.M., y Kelly C.T. Amylase, amyloglucosidase and related glucanases. Rose A.H. Economic Microbiology, Microbial Enzymes and Bioconversion, New York Academic Press Inc, vol. 5. Pp. 115-170 (1980).
- Wind R.D., Buitelaar R.M.G., Huizing H.J., and Dijkhuizen L. Characterization of a new Bacillus stearothermophilus isolate: a highly thermostable α-amylase producing strain. Appl Microbiol Biotechnol. 41:155-162 (1994).
- Brumm P.J., Hebeda R.E., and Teague W.M. Purification &caracterization of commercialized, cloned megaterium α-amylase. Part I: purification & hydrolytic properties. Starch. 43:319-323 (1991).
- Takasaki Y. Una amilasa productora de maltotriosa de subtilis. Agric Bio. Chem. 49:1091-1097 (1985).
- Lowry O.H., Rosebrough N.J., y Randall R.J. Protein measurement with the folin phenol reagents. J Biol Chem. 193: 265-275 (1951).
- Altschul S.F., Gish W., Miller W., Myers E.W., y Lipman D.J. Basic local alignment search tool. J Mol 215:403-410 (1990).
- Bhaskara R.K.V., Ashwini K., Gaurav K., and Karthik L. Optimization, production and partial purification of extracellular α-amylase from Bacillus marini. Arc Appl Sci Res. 3:33-42 (2011).
- GhoseK. Medición de las actividades de la celulasa. Pure Appl Chem. 59: 257-268 (1987).
- Haq U.I., Ashraf H., Iqbal J., y Qadeer M.A. Production of α-amylase by Bacillus licheniformis using an economical medium. Biores Technol. 87:57-61 (2003).
- Mishra S., and Behera N. Amylase activity of starch degrading bacteria is isolated from soil receiving kitchen wastes. Afr J Biotechnol. 7: 3326-3331 (2008).
- Fossi B.T., Taveaand F., and Ndjonenkeu T. Production and Partial Characterization of a Themostable amylase from Ascomycetes yeast strain isolated from starchy soils. Afr J Biotechnol. 4:14-18 (2005).
- Cordeiro C.A.M. Production and propertiesof α-amylase from thermophilic Bacillus sp. Brazil J Microbiol. 33:57-61 (2002).
- Asad W. Extracellular enzyme production by indigenous thermophilic bacteria: Purificación parcial y caracterización de α-amilasa por Bacillus sp. WA21. Pakistan J Botany. 43:1045-1052 (2011).
- Kim T.U. Purification and characterization of a maltotetraose-forming alkaline α-amylase from an alkalophilic Bacillus strain, GM8901. Appl Envin Microbiol. 61: 83105-3112 (1995).
- Sivaramakrishnan S., Gangadharan D., Madhavan K.N., y Pandey A. Solid culturing of Bacillus amyloliquefaciens for alpha amylase production. Food Technol 44:269-274 (2006).
- Abdel-Fattah Y.R., Soliman N.A., El-Toukhy N.M., El-Gendi H., and Ahmed R.S. Purification, and characterization of thermostable α-amylase produced by Bacillus licheniformis isolate AI20. J Chem. 2013:1-11 (2013).
- Thippeswamy S., Girigowda K., and Mulimani V.H. Isolation and identification of α-amylase producing Bacillus from Dhal
industry waste. Indian J Biochem Biophysics. 43: 295-298 (2006). - Abd-ElhalemT., El-Sawy M., Rawia F.G., and Khadiga A.A.T. Production of amylase from Bacillus amyloliquefaciens under submerged fermentation using some agro-industrialby-products. Ann Agri Sci. 60:193-202 (2015).
- Tamura K., and Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol Biol Evol. 10:512-526 (1993).

(Visitado 12.737 veces, 21 visitas hoy)